转录组如何研究噬菌体-细菌互作? 返回
研究背景
现阶段很多噬菌体-宿主关系研究都是基于大肠杆菌的,因此急需了解其他细菌噬菌体的入侵和裂解机理,拓展噬菌体-宿主关系研究,从而促进噬菌体治疗和发现抗细菌药物。φR1-37能够通过LPS上的外核多聚乙糖侵染多种结肠炎耶尔森杆菌(Yersinia enterocolitica),它的裂解周期大概40-50min,基因组大小262397bp,有367个潜在蛋白,是很好的研究材料。
研究策略
φR1-37侵染Y. enterocolitica 菌株YeO3-R1后,取2, 5, 10, 15, 21, 28, 35, 42, 49 min每个时间点(没有重复)进行有参转录组研究(细菌和噬菌体都有参考基因组)。

图1 文章思路
结果解读
1、室温下噬菌体明显感染细菌
在不同温度下(4、10、16、22和37℃),利用不同浓度(MOI)的φR1-37噬菌体感染细菌,发现不同浓度噬菌体在各个温度下都能感染宿主(图2)。研究同时发现,随着温度的升高,需要感染的噬菌体浓度和时间慢慢减少。但在37℃下,由于温度高,细菌变异较快,因此出现抗噬菌体现象(图2),这有可能限制后期实验采样。因此,综合来说,在室温(22℃)条件下,噬菌体侵染时间少,需要浓度低,而且抗噬菌体现象不明显,是作为后续噬菌体-细菌互作研究的理想培养条件。

图2 细菌在各温度下受侵染生存情况
2、噬菌体转录本在细菌大量表达
通过比较细菌体内的噬菌体转录组情况,发现在侵染后,细菌体内的噬菌体转录本占有率从3.27%(2min)升高到15.71%(50min)(图2),表示噬菌体入侵细菌之后,快速进入裂解过程。
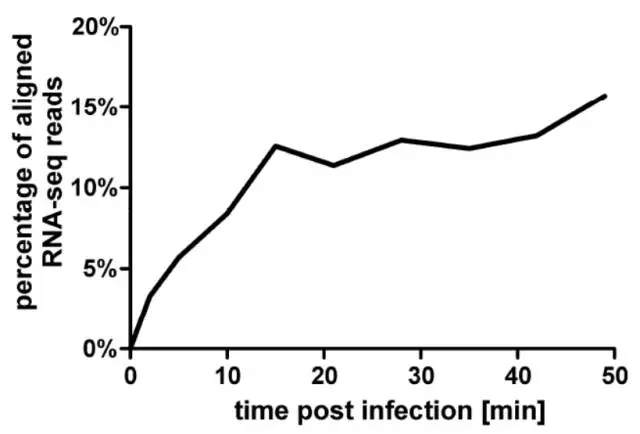
图2 不同时间细菌体内噬菌体表达情况
根据各个时间点样本的所有转录组情况(噬菌体和细菌)进行PCA分析,发现这些时间点可以明显地分为四个阶段(图3):感染前(也就是对照组,时间为0min);感染早期(2和5min);感染中期(10、15、21min);感染后期(28、35、42和49min)。这为后续趋势分析提供有力的分组信息。
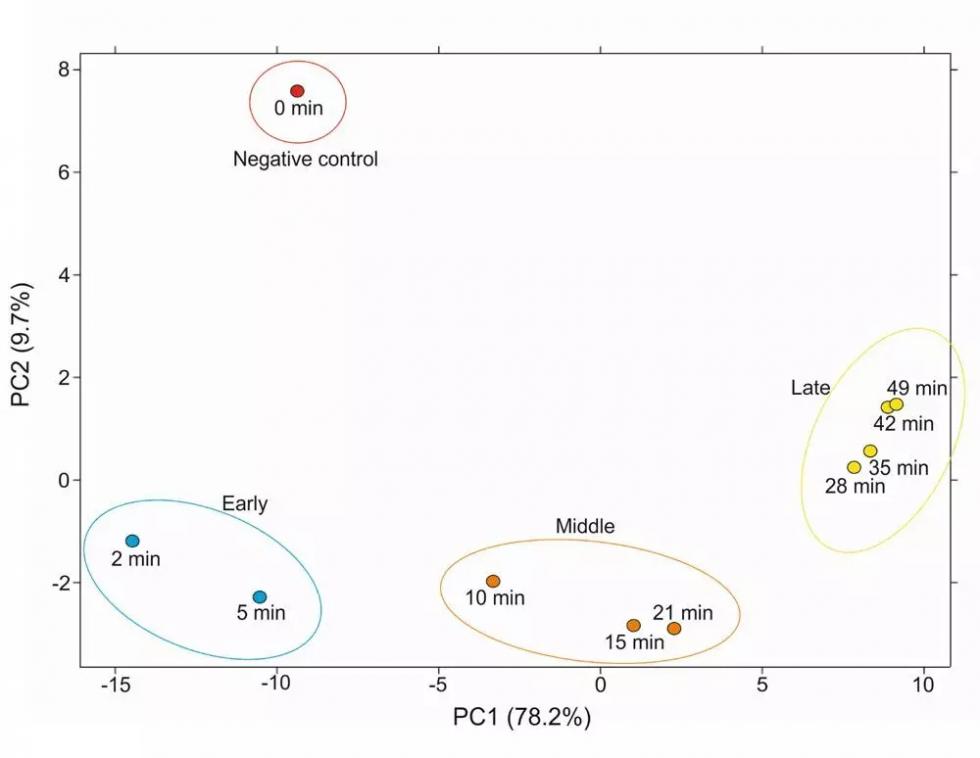
图3 不同时期样本PCA情况
3、差异基因趋势分析
根据4个时期的病毒基因表达情况来看,总体而言,各个时期基因表达的差异并不大,可认为比较类似,但依然存在部分差异基因。不同时期的差异基因基本可以分为四大类(图4):早期高表达(92基因)、中期高表达(8基因)、晚期高表达(94基因)和稳定表达(147基因)。这些不同时期高表达的基因包括病毒粒子结构蛋白、RNA结合蛋白和DNA修复蛋白等。
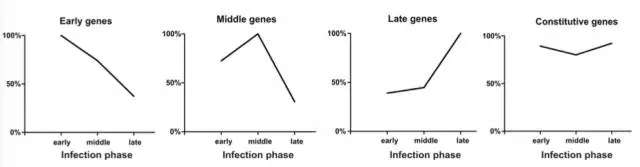
图4 病毒基因组在不同时期的表达模式
4、病毒RNA转录调控情况
作者研究完病毒转录组在不同时期的表达差异以及差异基因的功能后,进一步挖掘病毒的基因调控关系。其中转录组的调控元件预测(通过MEME)发现56个之前被实验证明的噬菌体启动子。这些启动子中有大量是中到弱水平的转录水平,这对应解释为什么在侵染过程中大量基因的表达并没有太大差异。另外,RNA测序还发现了10个新RNA(图5),其中9个预测能够参与基因调控。例如非编码RNA,misc_4可能调控宿主ptr 等三个蛋白。
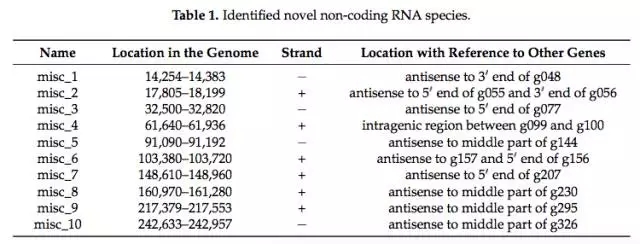
图5 新鉴定病毒非编码RNA
5、宿主对病毒感染的转录应答
研究完病毒的入侵机制后,转而研究宿主的侵染应答情况。通过比较早期(2-10min)和感染前(0min)、晚期(28-49min)和感染前、早期和晚期的宿主转录组变化情况(图6),发现:
早期 vs 0min:112个基因(占2.85%)在早期表达下调,只有7个上调,这些上调基因与离子转运等有关。
晚期 vs 0min:在侵染晚期,细菌的转录组变化更大,有129个差异基因上调,它们与ABC转运蛋白、膜蛋白以及应激蛋白、转录因子等有关。这些蛋白大多数与细菌应激相关。
早期 vs 晚期:细菌转录组的差异基因主要为上调(145/149)。
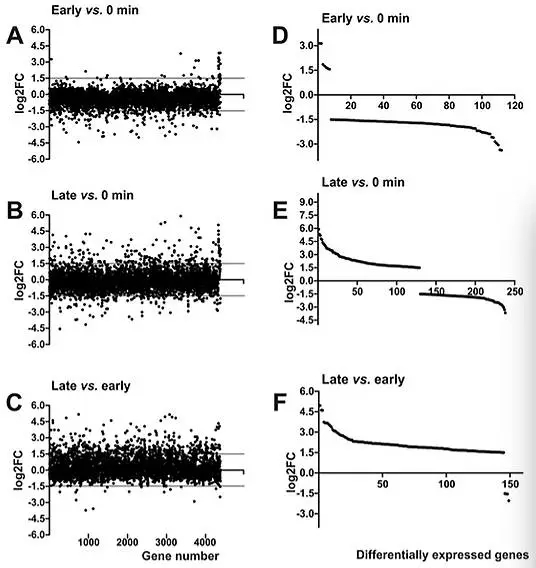
图6 不同时期的细菌转录组差异比较情况
总 结
利用转录组测序,本研究发现了噬菌体在侵染过程中的转录组变化情况,同时也了解了细菌在侵染过程中的转录应答反应。噬菌体在侵染的过程中,转录组变化不大;而细菌在侵染前期,基因表达主要以下调为主,但后期则大量基因上调表达。这些上调基因与多个应激行为有关。
在研究思路上,本文也有其精彩之处:
1、在确定需要进行趋势分析之前,作者并不确定其分组情况(采样时间),因此本研究采用密集采样策略,然后通过PCA分析进行明确分组后,最后很好地开展趋势分析研究。
2、本文给我们一个很好的研究思路:分散的采样信息,通过PCA进行分组整理,然后利用趋势分析和差异比较的方法,最后系统地找出样本规律。
3、最后,本文不但在入侵者(噬菌体)角度进行入侵机制研究,同时还从被侵染者(细菌)角度出发,完成整个侵染故事的叙述。
参考文献
【1】Leskinen K, Blasdel B G, Lavigne R, et al. RNA-sequencing reveals the progression of phage-host interactions between φR1-37 and Yersinia enterocolitica[J]. Viruses, 2016, 8(4): 111.
更多的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























