微生物群落功能预测的新方向 返回
微生物群落研究主要有16S/ITS多样性测序、宏基因组测序还有宏转录组测序等研究手段。其中16S/ITS等多样性测序由于价格便宜,性价比高而深受广大微生物研究者喜爱。
利用16S/ITS多样性测序,我们可以准确知道群落的物种结构,但越来越多的研究表明,微生物的群落功能组成比物种组成与环境关系更为密切[1],因此我们需要借助宏基因组等研究手段来获取群落功能信息。现阶段的宏基因组、宏转录组的价格相对较高,在经费有限的情况下,利用16S/ITS的测序结果进行功能预测是一个值得重视的方向。2013年所推出的PICRUSt就是一款常用的群落功能预测软件。
随着业务发展,我们基迪奥的生信团队也紧跟潮流发展,在最新的16S/ITS多样性测序报告中提供更多的功能预测服务,其中包括FUNGuild、Tax4fun、FAPROTAX、BugBase等软件的群落功能预测,下面我们来简单介绍一下这些软件的原理和作用:
1、Tax4Fun
Tax4Fun与PICRUSt类似,是基于16S的测序结果对群落进行宏基因组的功能预测,主要涉及的预测方向是KEGG类型的通路预测。官方说明表示Tax4Fun的预测结果比PICRUSt更为准确,这可能得益于Tax4Fun的预测是基于SILVA数据库,而PICRUSt是基于Greengenes数据库。Greengenes数据库自2009年开始已经没有继续更新,而SILVA数据库到目前为止还保持每月更新,因此物种信息更为全面。
同时,Tax4Fun的预测是基于已在KEGG上有注释的通路的OTU进行判断,而PICRUSt是基于祖先预测,在这些方面上,Tax4Fun的结果确实会更为“真实”。但不足的地方是,Tax4Fun并没有像PICRUSt的NSTI值类似的严格的筛选参数,因此其预测结果的真实性不好评估。因此,在PICRUSt的NSTI值>0.17时,建议两个软件的结果都可以作为参考。
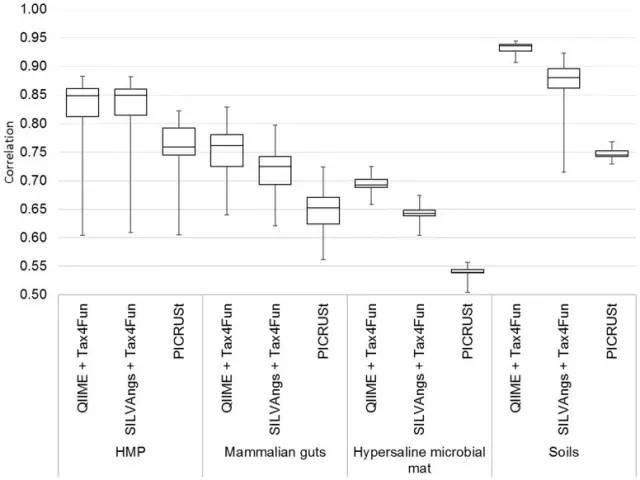
不同类型样本下的Tax4Fun与PICRUSt结果比较[2]
2、FUNGuild
FUNGuild是2016年所发表的一款基于ITS的功能预测软件。与其说FUNGuild是一种功能预测,我觉得它实际上更接近一种“表型”预测。与PICRUSt不同,由于真菌基因组数据的缺乏,很难走KEGG通路的预测路线。因此FUNGuild是基于已发表的文章数据所整合起来的一种称为“guild”的分类预测。Guild更类似是一种资源利用吸收所进行的功能分类,其中包括动物病原菌、植物病原菌、木质腐生菌等12类。通过对Guild的预测,可以从另外的生态功能角度研究真菌的功能。
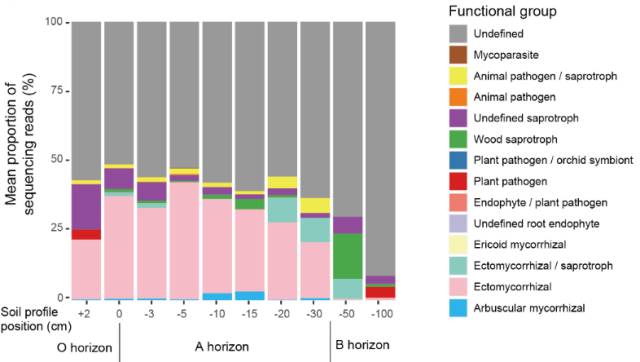
FUNGuild预测结果[3]
3、FAPROTAX
FAPROTAX是一款在2016年发表在SCIENCE上的较新的基于16S测序的功能预测软件。它整合了多个已发表的可培养菌文章的原核功能数据库,数据库包含超过4600个物种的7600多个功能注释信息,这些信息共分为nitrate respiration, methanogenesis, fermentation 和plant pathogenesis等80多个功能分组。如果PICRUSt在肠道微生物研究更为适合,那么FAPROTAX尤其适用于生态环境研究,特别是地球化学物质循环分析。
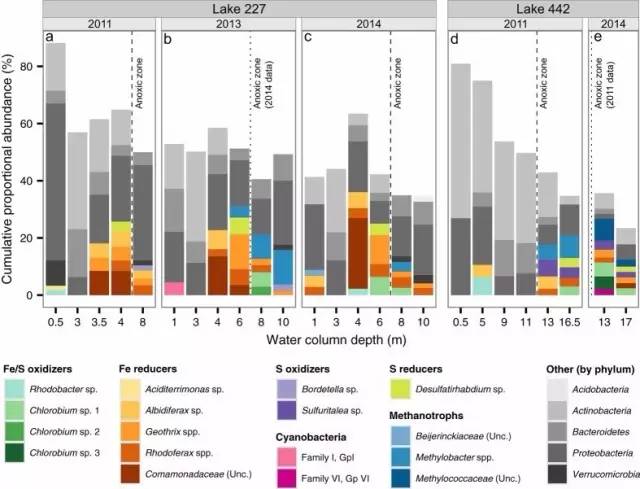
FAPROTAX结果[4]
4、BugBase
Bugbase也是16年所提供服务的一款免费在线16S功能预测工具,到今年才发表文章公布其软件原理。该工具主要进行表型预测,其中表型类型包括革兰氏阳性(Gram Positive)、革兰氏阴性(Gram Negative)、生物膜形成(Biofilm Forming)、致病性(Pathogenic)、移动元件(Mobile Element Containing)、氧需求(Oxygen Utilizing,包括Aerobic、Anaerobic、facultatively anaerobic)及氧化胁迫耐受(Oxidative Stress Tolerant)等7类。BugBase比其他软件的一个好处就是,它不单只可以进行功能的预测,如果同时输入注释物种的tag信息,还可以针对不同表型进行统计图表展示。

ugbase不同表型下的物种统计结果[5]
总 结
这几款软件针对不同数据库和marker基因,能够最大效率地利用多样性测序数据进行一定的功能分析,这是一个极具性价比的分析手段。同时,不同软件还可以针对不数据库进行预测,能够在不同层面进行研究。如果有兴趣的客户,可以联系我们开展相关的分析业务。
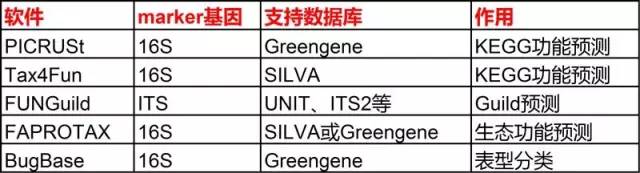
表1.各功能预测软件比较
[1] Gibbons S M. Microbial community ecology: Function over phylogeny[J]. NatureEcology & Evolution, 2017, 1: 0032.
[2] Aßhauer K P, Wemheuer B, Daniel R, et al. Tax4Fun: predicting functional profiles from metagenomic 16S rRNA data[J]. Bioinformatics, 2015, 31(17): 2882-2884.
[3] Toju H, Kishida O, Katayama N, et al. Networks Depicting the Fine-Scale Co-Occurrences of Fungi in Soil Horizons[J]. PloS one, 2016, 11(11): e0165987.
[4] Schiff S L, Tsuji J M, Wu L, et al. Millions of Boreal Shield Lakes can be used to Probe Archaean Ocean Biogeochemistry[J]. Scientific Reports, 2017, 7.
[5] Thomas A M, Jesus E C, Lopes A, et al. Tissue-associated bacterial alterations in rectal carcinoma patients revealed by 16S rRNA community profiling[J]. Frontiers in Cellular and Infection Microbiology, 2016, 6.
更多的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























