蓖麻籽lnc-RNAs表达网络与遗传模式的差异分析
|
合作单位:中科院昆明植物研究所
发表期刊:The Plant Journal
影响因子IF:5.901
|
研究背景
|
蓖麻是非常重要的经济作物,此前作者与基迪奥合作已经收获了两篇高分文章,文章1进行了蓖麻籽基因组印记的研究(IF:8.8),文章2 进行了胚与胚乳甲基化差异机制的研究(IF:6.8)。
这一次作者紧跟lncRNAs的研究热点,首先从lncRNAs差异表达及功能特征揭示其对胚与胚乳发育的调控,然后将文章1、文章2研究思路应用于lncRNAs的研究,从甲基化和基因组印记角度进一步揭示lncRNAs的差异机制。
该系列研究从2013年启动以来,基迪奥生物长期持续地为合作方提供个性化生物信息服务,有力辅助了该项目持续产出三篇文章。
|
研究思路
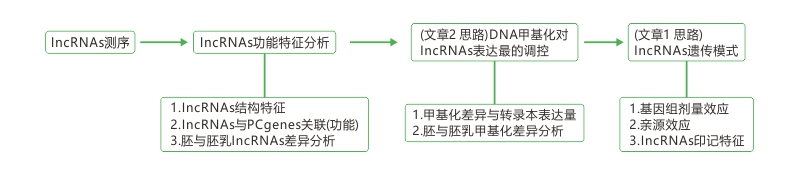
图1.研究思路
研究结果
| 1.lncRNAs功能分析
1.1 lncRNAs特征
10个样本序列组装过滤后获得5356lncRNAs,平均长度为803 nt。与拟南芥、水稻、等已知lncRNAs相比,蓖麻籽lncRNAs序列保守性极低,但位置保守性较高。16.3% lncRNAs与转座子序列重叠(TE-lncRNAs)。
|
|
1.lncRNAs功能分析
1.1 lncRNAs特征
10个样本序列组装过滤后获得5356lncRNAs,平均长度为803 nt。与拟南芥、水稻、等已知lncRNAs相比,蓖麻籽lncRNAs序列保守性极低,但位置保守性较高。16.3% lncRNAs与转座子序列重叠(TE-lncRNAs)。
1.2 lncRNAs与PCgenes关联
胚、胚乳组织中,lncRNAs的表达量显著低于PCgenes的表达量。作者鉴定了顺式(500 bp < 距离 < 5kb)和反式(距离 > 5 kb)lncRNAs与PCgenes、PCgenes启动子的pair(图2c),相关性分析呈现了较强的正相关(图2d、2e)。
|
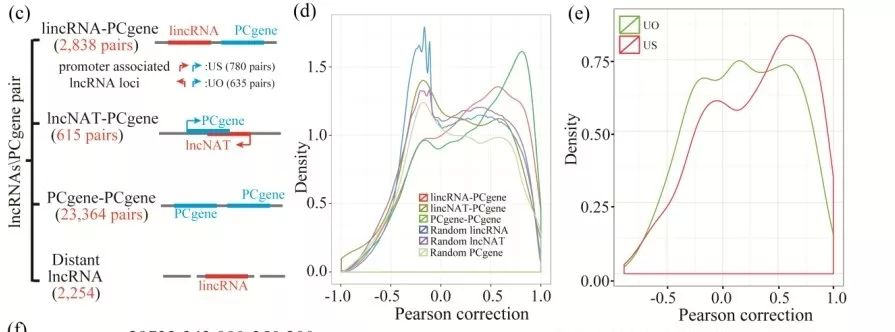
图2 lncRNAs与PCgenes pair数目和相关性
|
|
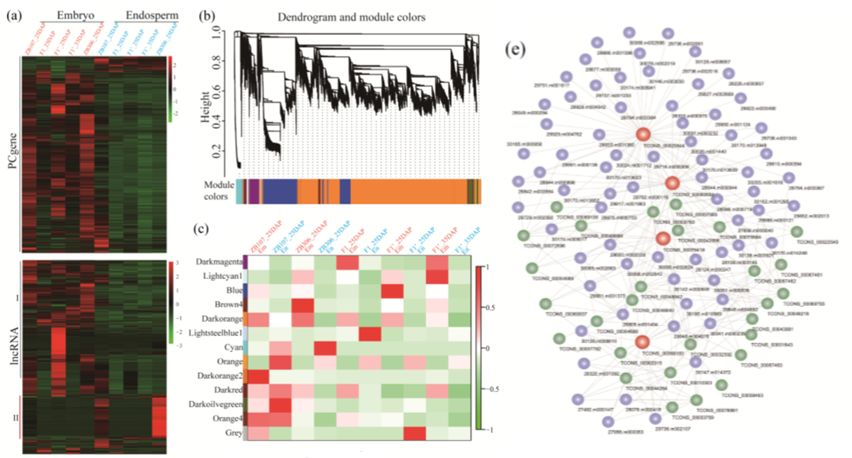
图3 胚、胚乳中转录本(lncRNAs和PCgenes)共表达分析
a.转录本基因表达量热图 b.WGCNA分析的层次聚类树 c. 模块与组织特征的相关性
e.darkorange模块的部分网络图,蓝色与绿色分别表示lncRNAs和PCgenes
|
1.3 胚、胚乳转录本表达量差异
相对于PCgenes,lncRNAs的组织特异性更高(图3a),暗示胚、胚乳的发育差异源于转录本的表达量差异,尤其是lncRNAs。权重基因共表达网络分析(WGCNA)获得13个模块(图3b),其中2个模块(图3c)与胚和胚乳的关联最显著:
1)模块darkorange含在胚中高表达的转录本,主要为核糖体合成组装,RNA剪切、转运、降解等代谢,4种核心lncRNAs(图3e)关联的PCgenes与调控胚发育的转录因子相关;
2)模块orange含在胚乳中高表达的转录本,主要为碳、氮、氨基酸、黄铜合成等代谢,核心lncRNAs关联的PCgenes与糖酵解相关。
|
|
2.DNA甲基化对lncRNAs的调控
除了从lncRNAs功能角度分析样本差异,作者还从lncRNAs被调控的角度更深入揭示上游DNA甲基化对样本差异的影响。
胚乳中lncRNAs与PCgenes的 CG和CHG甲基化水平都低于胚,但胚乳和胚中CHH甲基化水平相近(图4a)。比较lncRNAs及其上下游2kb区域内甲基化水平与表达量的关系,作者发现lncRNAs表达量与三种甲基化水平都呈负相关(图4b)。同时,胚体外实验证明,去甲基化处理后84.4%的lncRNAs表达量上调(图4c)。
|
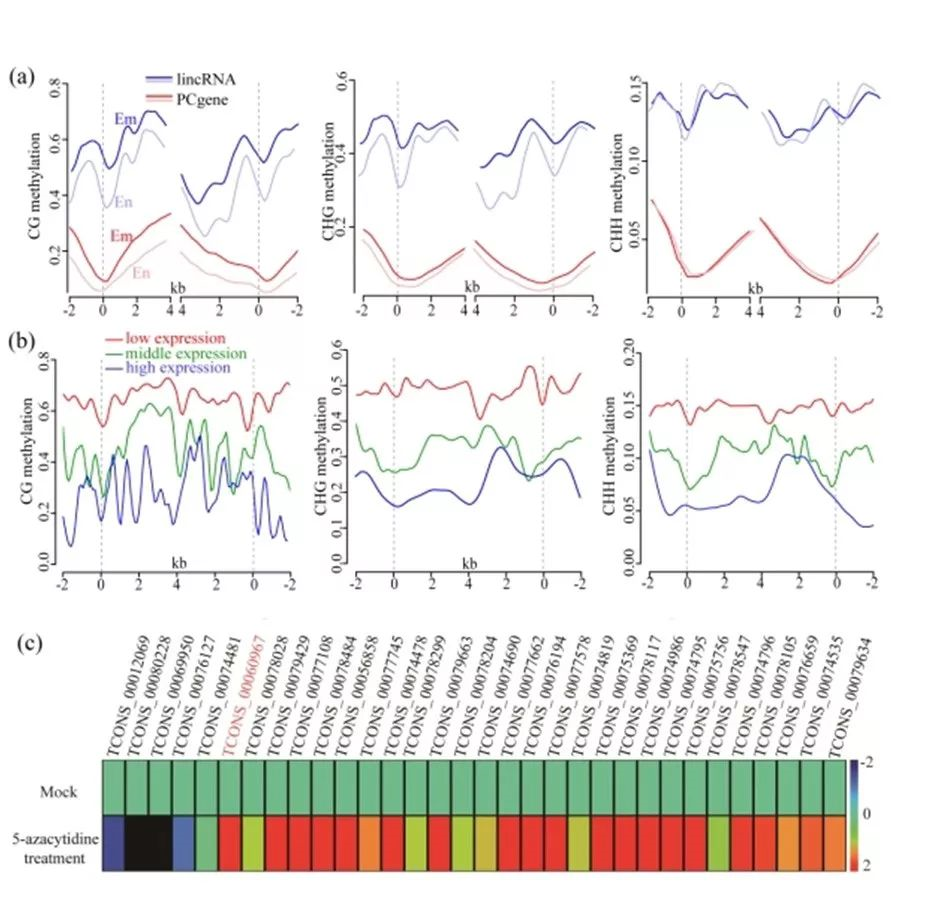
图4 a.胚、胚乳中转录本(lncRNAs和PCgenes)甲基化水平分析(En, 胚乳;Em,胚)
b. lncRNAs表达量与甲基化水平对比 c.胚体外甲基化实验lncRNAs表达量
|
|
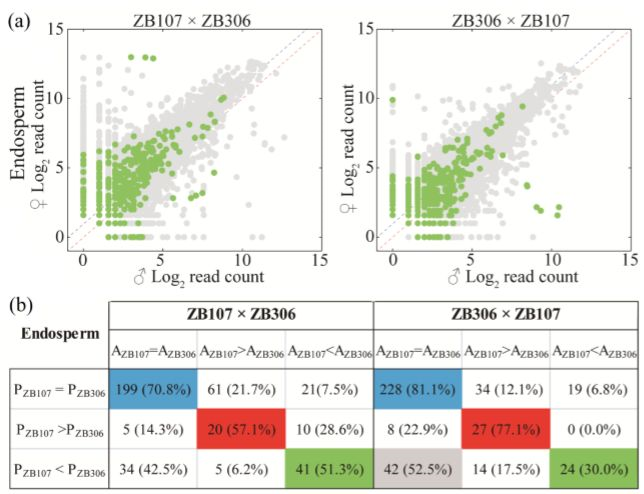
图5 不同杂交系胚乳中等位lncRNAs的表达量
|
3.lncRNAs的遗传模式
除了被DNA甲基化影响,作者认为lncRNAs的表达也有自己的遗传模式,如基因组剂量效应、亲源效应等。
3.1 基因组数量与来源对lncRNAs的影响
基于SNPs分子标记统计不同亲本来源lncRNAs表达量,结果显示,胚乳中,部分符合预期母本与父本2:1的比例(因为胚乳为3倍体,含母本基因组2份,父本1份)(图5a),暗示基因组剂量效应对lncRNAs表达量有影响。对于小部分的偏差,作者补充了亲源效应分析(图5b),发现当亲本中lncRNA表达量差异很大时,就会影响子代中母本与父本来源lncRNAs表达量的比例。
|
|
3.2 印记lncRNAs的鉴定与聚类
研究获得20个母本印记lncRNAs,以及新的56个亲本印记PCgenes。4种 lncRNAs验证实验显示,
3个符合预期的母本印记,1个表现出不完全印记(图6)。
|
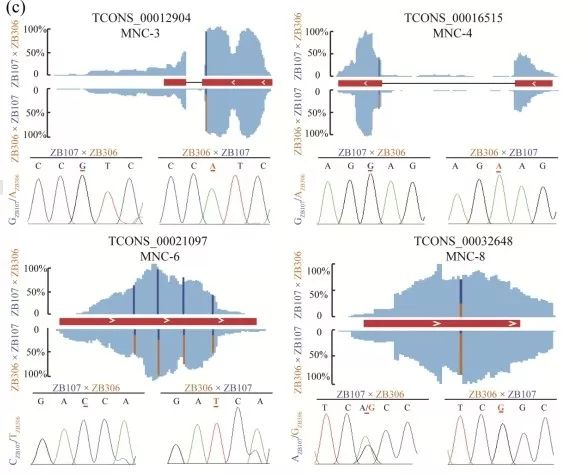
图6 印记lncRNAs的表达量验证
|
小结
|
作者充分利用前两篇文章中甲基化与基因组印记的成果和经验,在此篇文章中结合lncRNAs数据进行了全面系统的差异分析,使生物学问题的研究更加充分深入,获得主要成果如下:
1. 从lncRNAs丰度和功能角度初步揭示了造成胚、胚乳差异发育的原因;
2. 从DNA甲基化水平初步揭示了lncRNAs差异表达的原因;
3. 从基因组剂量效应、亲源效应、基因组印记方面揭示了lncRNAs的遗传模式。
|
|
参考文献
Xu W, Yang T, Wang B, et al. Differentialexpression networks and inheritance patterns of long non‐coding RNA s in castorbean seeds[J]. The Plant Journal, 2018.
|