|
靶向代谢组学(Targeted metabolomics)针对特定某一物质或某一类代谢物进行检测的代谢组方法,具有特异性强、检测灵敏度高和定量准确等优点。利用AB SCIEX 5500 QQQ UPLC-MS液质联用仪可检测到含量更低代谢物,应用范围更广。通过与标准品的比对分析等获得目标代谢物的绝对丰度信息。随着分析研究的逐步深入,代谢网络逐渐全面系统,生物学研究趋于具体代谢通路的细节探索。靶向代谢组学即在精细化的代谢组学研究中发挥着重要作用。 |
应用领域
|
植物代谢组:用于植物主要和次生代谢途径、植物病虫抗性机理、植物微生物相互作用等研究;
食品代谢组:用于食品营养功能评估、食品安全评估等;
临床代谢组:揭示疾病的分子机理,寻找早期诊断的血液和尿液标记物,肠道和口腔代谢等。
靶向代谢化合物检测部分列表:
|
技术路线
分析内容
| 标准信息分析 | 定制化信息分析 |
|
a) 物质标准曲线绘制 b) 原始数据预处理 c) 物质绝对定量 |
a) 多组学数据关联分析 |
|
样品要求/项目周期
请咨询当地销售或拨打电话:020-84889324(医学)、020-84889314(农学)了解详情。 |
参考文献
|
[1].Wang, Rui, et al. "Identification of novel pathways for biodegradation of bisphenol A by the green alga Desmodesmus sp. WR1, combined with mechanistic analysis at the transcriptome level." Chemical Engineering Journal.321 (2017): 424-431.
[2].Tieman D, Zhu G, Resende M F R, et al. A chemical genetic roadmap to improved tomato flavor[J]. Science, 2017, 355(6323): 391-394.
[3].Yu C, Zhao X, Qi G, et al. Integrated analysis of transcriptome and metabolites reveals an essential role of metabolic flux in starch accumulation under nitrogen starvation in duckweed[J]. Biotechnology for Biofuels, 2017, 10(1): 167.
[4].Liu K, Feng S, Pan Y, et al. Transcriptome Analysis and Identification of Genes Associated with Floral Transition and Flower Development in Sugar Apple (Annona squamosa L.)[J]. Frontiers in Plant Science, 2016, 7.
|
|
代谢工程提高微藻产油量
|
合作单位:暨南大学 发表期刊:《Metabolic Engineering》 影响因子IF: 8.142 |
研究背景
|
三角褐指藻,参考基因组已经公布。基于基因组信息,研究者非常方便了解脂类代谢通路和关键调控基因,从而为藻类的精准改良提供了可能。因为脂类物质是还原产物,所以其从头合成需要持续消耗机体中的NADPH(还原型辅酶II)来提供还原力,将基团(CH3-CO-)还原为脂肪酸的酰基链(-CH2-CH2-)。但微藻产油的遗传和生化机制还未被完全揭示。 本研究将调控的重点放在磷酸戊糖途径(PPP),PPP可以产生NADPH和磷酸戊糖,在动物中可以提供肝脏中50%-75%的还原当量。PPP在NADPH合成和维持机体氧化还原平衡起着非常关键的作用。而G6PD是催化PPP途径中的上游基因,且在各个物种都已经被报道,是脂类合成的关键基因。因此,作者就以这个基因为操纵目标,最终显著提高了三角褐指藻的产油量。 |
研究结果
|
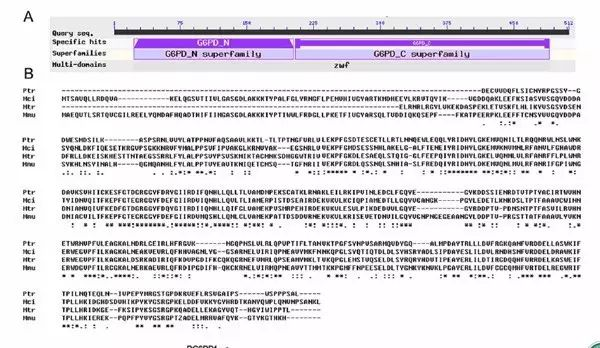
图1 prG6PD的序列特性解析 |
1.目标基因的序列分析和转基因构建微藻工程株
首先作者对三角褐指藻G6PD(ptG6PD)进行了序列分析。目标序列的分析,在组学类文章里面经常会出现。例如,在植物DNA甲基化研究中,通常会分析该物种DNA甲基化相关酶的序列特性;在植物抗病研究中,会分析抗病基因(R基因)的序列结构特点等。在分析中,最常见的分析条目包括:结构域分析、多物种保守性分析以及基因进化关系分析。 比如在这篇文章正文中,作者分析了ptG6PD基因的结构域(图1A)和保守性。从中我们可以看出G6PD有两个结构域,其动植物种中非常保守。该基因如此保守,也间接暗示了其在脂类代谢中起着不可替代的作用。 备注:图B中的4个物种分别为三角褐指藻(ptr)、毛霉菌(Mci)、紫花苜蓿(Mtr)、小鼠(Mmu)。
|
|
然后作者构建prG6PD的过表达载体到三角褐指藻中,发现该基因的表达量和酶活都显著提高了——工程株构建成功。
脂肪酸成分的变化 通过气相色谱-质谱分析(GC-MS)分析,发现了基因工程株生产的脂肪酸成分发生了明显的变化。总饱和脂肪酸(SUM SFA)略微提高,其中C16:0占的比例最大。总单不饱和脂肪酸(SUM MUFA)提高了58.5%,包括C16:1,C18:1提高明显。但总多不饱和脂肪酸(SUM PUFA)在基因工程株中降低了22.7%。
|
图2 prG5PD表达量提高了2.4 和2.7倍(A) |
图3 脂肪酸成分的变化 |
|
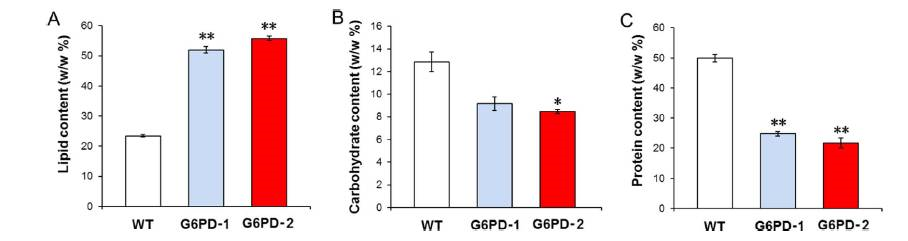
图4 初生代谢物的含量变化 |
2. 初生代谢物的含量也发生了变化(图4) 3. 蛋白质谱和代谢组揭示基因工程微藻的代谢转化机理 通过iTraq蛋白质谱分析,发现上调的通路包括:脂肪酸合成和代谢、PPP(磷酸戊糖途径)、TCA(三羧酸循环)、粘多糖代谢、能量代谢和氨基酸代谢等。同时,发生下调的通路包括:脂肪酸延伸、碳水化物代谢、核酸代谢、尿素循环和维生素循环。这些上调的通路有利于更多的碳流向脂肪酸合成以及脂类的累积。
而G6PD启动了磷酸戊糖途径的第一步,同时可以生成更多的NADPH用于脂类合成。代谢组分析也得出了与蛋白质谱相似的结论,包括PPP、糖酵解和TCA相关的代谢物发生显著的提升。 |
|
参考文献 [1]Jiao Xue,Srinivasan Balamurugan,Da-Wei Li,Yu-Hong Liu,Hao Zeng,Lan Wang,Wei-Dong Yang,Jie-Sheng Liu,Hong-Ye Li. Glucose-6-phosphate dehydrogenase as a target for highly efficient fatty acid biosynthesis in microalgae by enhancing NADPH supply[J]. Metabolic Engineering,2017,41. |