PNAS文献解读:大麦R基因导入拟南芥中的抗病研究 返回

文章主体思路

文章中的细节
首先我们可以看到:
1、尽管是一篇PNAS文章,但文章的主体结构非常简单(三个小节)。但其实在细节逻辑的论证上非常严谨。
2、RNA-seq在这里只是一个工具,帮助支撑文章的一个论点(大麦MLA的靶基因在物种间是保守的);
1、总体设计---------------------典型的2X2X4的三因素实验,每个条件设置3个重复,一共48 样本。
三个条件分别是:
1)不同的植株,分别为MLA 转入/未转入的植株
2)不同的病原真菌处理,抗病基因MLA只对其中一种具有抗性;
3) 感染后的4个时间点,分别为6h,12h,18h,24h。
2、设计的小结:
1)对照非常重要。任何实验,没有对照就没有充足的说服力。而本文,在宿主和病原水平都设置了两种极端的参照,从而更具说服力。
2)时间节点。抗病应答,涉及早期、中期、晚期应答,不能只检测一个时间点,否则可能会损失关键信息。
3)实验重复。虽然,我们有丰富经验对付审稿人拷问“你的项目怎么没有重复”。但在经费许可的情况下,还是要设置一定的重复才算严谨。因为任何实验都是存在误差的,因此没有实验重复而得出的结论是不严谨的。
1、这个研究的实验设计是2X2X4的三因素(植物品系、病原株系、处理时间)设计。由于设计较为复杂,如何进行差异表达分析成为了难点。
2、传统的两两样本组差异分析,或简单的趋势分析并不足够。所以作者使用了多元回归的策略,找到不同病原应答出现差异的基因。
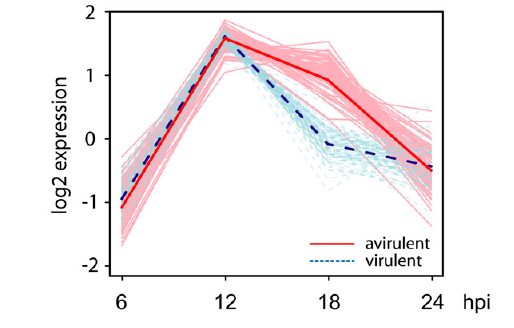
如图,87个对不同病原株系(有毒和无毒组)应答不同的基因。红色线条是这些基因在无毒组(有抗性反应)的表达模式,蓝线是这些基因在有毒组(无抗性反应)的表达模式。
我们可以明显看到,这些基因主要在18h出现最大的差异。
而其中44个基因的GO注释是“response to chitin”(对真菌细胞壁的应答有关)
文章告诉我们什么?
1、植物抗病基因的作用,在种间具有很高的保守性。
在抗病基因功能验证方面,非模式物种的抗病基因,在模式物种中也可能得到验证。
2、抗病基因研究中上游比较重要。
不管通路上游如何识别和应答病原蛋白,信号的下游通路都是类似的。所以,我们要研究不同品系抗病的能力和机理,把重点放在早期应答上,或许更有意义。
3、测序只是工具,而不是项目的全部。
从整个行业的发展趋势来说,测序越来越成为一个工具,而不是研究的全部(除了基因组测序、大规模重测序这样的典型测序类文章)。
关于植物抗病研究RNA-seq结果的解读,涉及到早期、应答相关基因的文章解读,可点击下面链接
文章链接:http://www.genedenovo.com/news/250.html
更多基迪奥精彩原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























