环状RNA编码的小肽通过调节STAT1的SUMO化与磷酸化的平衡来阻断免疫原性铁死亡
|
合作单位:中国药科大学
发表期刊:Molecular Cancer
|
| 随着2017年的第一篇报道环状RNA被发现可翻译, 环状RNA翻译为环状RNA的功能研究提供了新的方向。从逻辑上说,多肽属于微量就可以起到巨大效应的分子,所以对于较低丰度的环状RNA更有可能通过翻译多肽来起到生物学作用。中山大学附属第一医院的张弩教授在之前的研究中,分别报道了蛋白编码基因(FBXW7和SHPRH)在形成环状RNA后形成新的功能多肽,且具有肿瘤抑制功能。 |
|
免疫检查点阻断(ICB)疗法是目前黑色素瘤的主要治疗方法,但其临床响应率仍然较低,需要寻找新的治疗策略。而CircRNAs往往在ICB治疗的癌症中持续高表达,这些CircRNA可以影响肿瘤免疫微环境,并且与肿瘤免疫治疗的疗效相关。
结合RNA-seq、Ribo-seq和质谱技术,探索影响黑色素瘤ICB疗效的因素,并找到可能的circRNA治疗靶点。
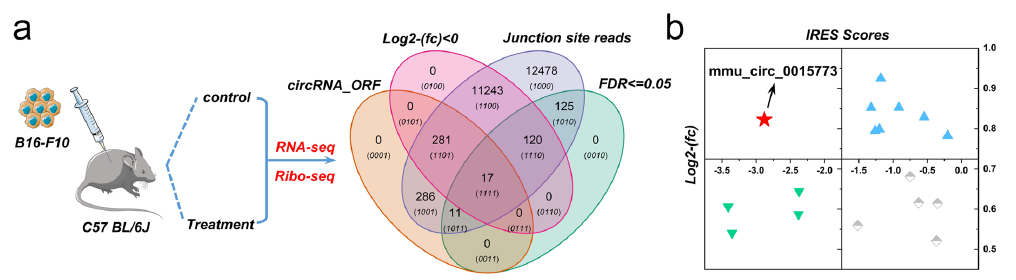
经过PD-1抑制剂治疗和脂质体药物与PD-1联合抑制剂治疗小鼠的黑色素瘤肿瘤组织
1.基于RNA-seq和Ribo-seq寻找目标circRNA
为找到在ICB治疗中持续高表达的CircRNAs,作者对单独使用PD-1抑制剂和与脂质体药物联合治疗的小鼠黑色素瘤组织进行了RNA-seq和Ribo-seq,通过差异分析,作者筛选出了17个在单独治疗中上调,而在联合治疗中下调的CircRNA(图1a)。接着,作者使用IRESfinder软件评估了这17个circRNAs的内部核糖体进入位点(IRES)区域活性,并联合差异分析(图1b)发现,与单一治疗相比,联合治疗后circPIAS1表达下调,表明其可能是促进黑色素瘤生长和阻碍免疫药物的有效性的关键circRNA。(基于Ribo-seq来鉴定circRNA的方法可见文末的总结处)
图1 a.通过RNA-seq和Ribo-seq筛选到17个候选circRNA;b.使用IRES评分和差异指数进一步鉴定高蛋白编码潜力的circRNA
作者通过分析circRNA的序列特征,发现其来源于PIAS1基因的外显子3-6,且小鼠和人的circPIAS1有94.71%的序列保守性(图1c-d)。此外,通过gDNA和cDNA的PCR扩增,证实了circPIAS1在人和小鼠黑色素瘤细胞中确实存在。通过RNaseR耐受性实验,进一步确认了circPIAS1的环状结构。通过FISH实验,发现circPIAS1主要定位于细胞核内,且与正常组织相比,黑色素瘤组织中circPIAS1的表达水平显著升高。
通过circPrimer2.0数据库预测,作者发现circPIAS1可能编码一个108aa长度的多肽circPIAS1-108aa(图2a)。通过双荧光素酶报告实验,作者确定了小鼠和人circPIAS1的IRES序列分别位于81-254bp和21-194bp处,且通过截断IRES序列,发现主要活性区域集中在后半部分。最后,通过LC-MS/MS检测,作者成功鉴定了circPIAS1-108aa的多肽片段,包括其特异的尾部序列,确定了circPIAS1在黑色素瘤细胞中的编码能力。
此外,作者还评估了circPIAS1过表达和敲低对circPIAS1-108aa表达水平的影响,结果显示circPIAS1过表达可显著增加circPIAS1-108aa的表达,而敲低则相反。这些结果表明,circPIAS1确实存在与黑色素瘤细胞中且能编码一个108aa长度的多肽。
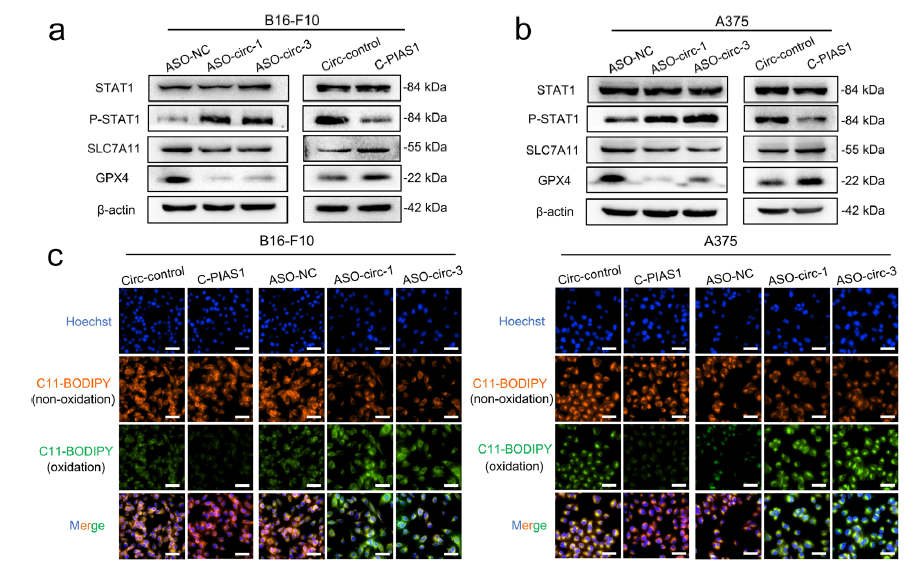
首先通过CCK-8和EdU等一系列实验检测过表达circPIAS1对黑色素瘤细胞增殖的影响,结果发现circPIAS1的促癌作用依赖于其编码的108aa肽,circPIAS1-108aa蛋白水平的升高显著促进了黑色素瘤细胞的增殖。进一步的circPIAS1体内敲低和过表达实验发现,CircPIAS1-108aa可通过抑制STAT1磷酸化来激活SLC7A11/GPX4信号通路,从而促进肿瘤生长和逃避免疫治疗。
图3.CircPIAS1-108aa通过抑制P-STAT1/SLC7A11/GPX4信号通路保护黑色素瘤免受铁死亡。
5.CircPIAS1-108aa降低ICB治疗的有效性的机制
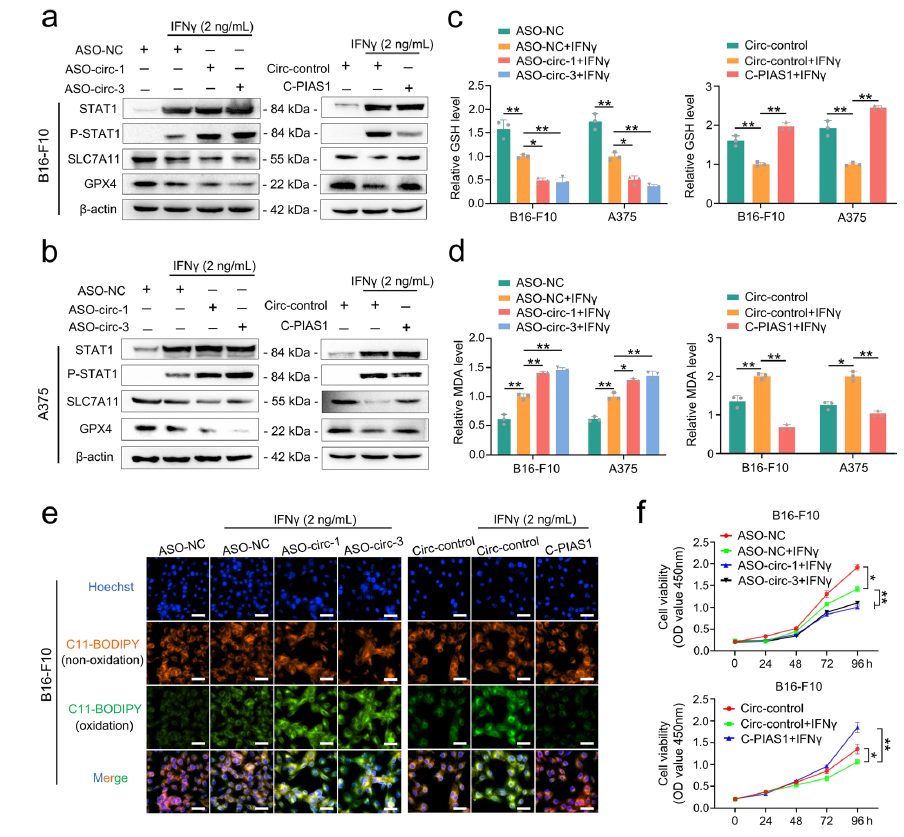
研究表明,用ICB治疗后,活化的CD8T细胞会产生高水平的IFNγ进而促进STAT1磷酸化,抑制SLC7A11/GPX4信号通路,并诱导黑素瘤中的铁死亡。因此,作者推测circPIAS1-108aa可能通过抑制这种特异性通路来抵消IFNγ的影响。通过检测IFNγ诱导下circPIAS1敲低或过表达对P-STAT1/SLC7A11/GPX4信号通路、GSH和MDA水平以及细胞膜脂质过氧化损伤的影响发现,circPIAS1-108aa确实可通过抑制STAT1磷酸化来抵抗IFNγ诱导的免疫性铁死亡,这反过来降低了ICB治疗的有效性。
图4.CircPIAS1-108aa通过抑制STAT1磷酸化抵抗IFNγ诱导的免疫原性铁死亡
6.circPIAS1-108aa抑制STAT1磷酸化的具体机制
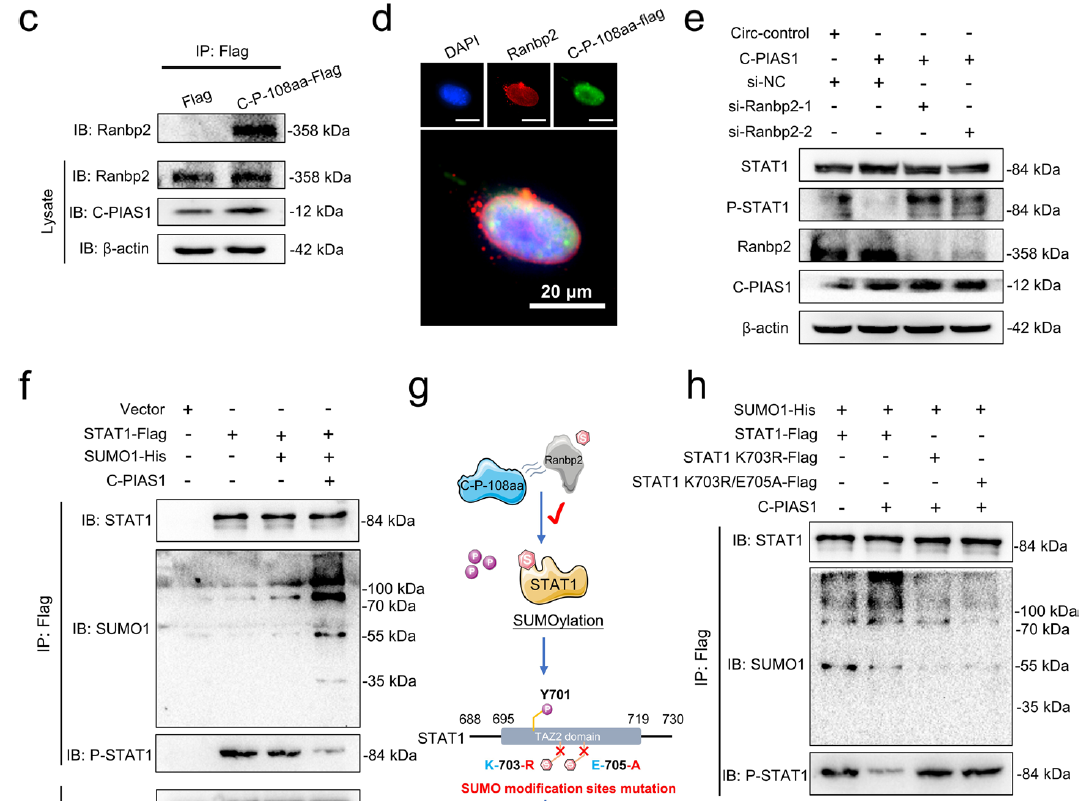
通过circPIAS1过表达、敲低和敲除实验来观察STAT1磷酸化水平的变化,作者发现circPIAS1-108aa对STAT1磷酸化的抑制作用是独立的,不依赖于pias1。因此,作者又通过构建STAT1-flag、SUMO1-His和circPIAS1过表达载体,并使用IP和WesternBlot检测STAT1的SUMO化和磷酸化水平,最终发现circPIAS1-108aa通过促进STAT1的SUMO化修饰来抑制其Tyr701位点的磷酸化,从而影响下游信号通路,最终抵抗免疫性铁死亡。
图5.circPIAS1通过招募Ranbp2增强STAT1的SUMO化修饰来抑制STAT1的磷酸化
7.抑制circPIAS1可以显著增强PD-1抑制剂的抗黑色素瘤效果
以上研究均在体外进行,接下来作者为确定circPIAS1在黑色素瘤中的作用及其作为治疗靶点的潜力,进行了一系列体内实验。
作者在C57BL/6J小鼠中建立了黑色素瘤皮下接种模型,并分成四组给予不同的治疗,通过观察小鼠体重、脏器健康、肿瘤体积、P-STAT1水平、铁死亡和免疫微环境等来评估疗效,结果发现抑制circPIAS1可以显著增强PD-1抑制剂的抗黑色素瘤效果,这为提高黑色素瘤免疫治疗效果提供了新的思路。
在这篇文章中,作者巧妙地利用RNA-seq、Ribo-seq和质谱技术,对黑色素瘤肿瘤组织进行分析,并发现了circPIAS1这个在黑色素瘤中高表达且具有编码蛋白的潜力的circRNA。后续通过一系列实验发现,circPIAS1是限制黑色素瘤免疫治疗效果的关键因素。抑制circPIAS1可以增强PD-1抑制剂的抗黑色素瘤作用,为提高ICB治疗效果提供了新的策略。
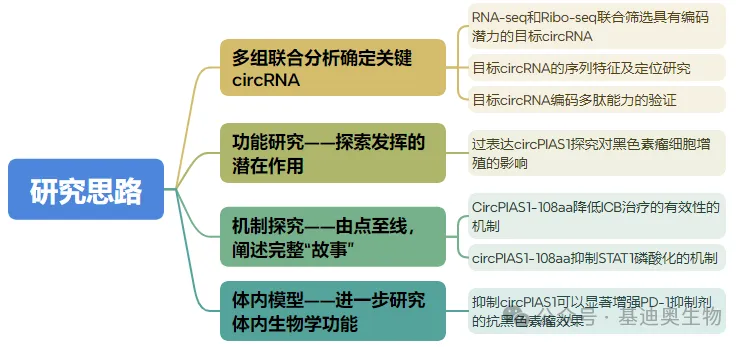
该文章先在体外完成了可编码circRNA的确定+小肽的功能研究+机制探究,接着又构建了小鼠的动物模型,进行了体内实验的功能探究,文章内容十分完整和丰富,有想研究circRNA编码小肽能力研究小伙伴可以借鉴一下此文章的研究思路(图8)。
|
|
参考文献:
Zang X, He X Y, **ao C M, et al. Circular RNA-encoded oncogenic PIAS1 variant blocks immunogenic ferroptosis by modulating the balance between SUMOylation and phosphorylation of STAT1[J]. Molecular Cancer, 2024, 23(1): 207.
|