| 外显子组是一个物种基因组中所有外显子区域的总和。外显子组测序是指利用序列捕获技术对全基因组外显子区域DNA进行富集再进行高通量测序的方法。该方法能够获得指定外显子捕获平台探针设计区域及侧翼200bp序列的遗传信息,极大的提高了人类基因组中外显子区域的研究效率,显著降低了研究成本。主要用于识别和研究与疾病、种群进化相关的编码区域内的结构变异。结合大量的公共数据库提供的外显子数据,有利于更好地解释所得变异结构之间的关联和致病机理。 |
应用领域
|
1. 已知候选位点筛选及验证;
2. 稀有或新变异位点检测;
3. 个性化诊断、药物靶点筛选。
|
技术路线
分析内容
 |
|
样品要求/项目周期
请咨询当地销售或拨打电话:020-84889324(医学)、020-84889314(农学)了解详情。 |
|
参考文献 [1] Xiong M, Heruth D P, Chaudhary S, et al. Identification Of Novel Single Nucleotide Polymorphisms Associated With Acute Respiratory Distress Syndrome By Meta-Analysis Of Exome-Seq Data[M]//C21. OMICS IN LUNG DISEASE. American Thoracic Society, 2017: A4976-A4976.
[2] Yang M, Xiong M, Chen H, et al. Novel Genetic Variants Associated with Child Refractory Esophageal Stricture with Food Allergy by Exome Sequencing[J]. Nutrients, 2017, 9(4): 390.
[3] Sant D W, Tao W, Field M G, et al. Whole exome sequencing of lacrimal gland adenoid cystic carcinoma[J]. Investigative ophthalmology & visual science, 2017, 58(6): BIO240-BIO246.
|
|
Q1: 全外显子组测序必须有参考基因组吗?
A: 必须有,如果没有参考因组,要提供近缘物种的序列,但不能保证捕获结果的可靠性。因为捕获探针是根据提供的参考序列来设计的,如果已知目标区域与参考基因组比有较大出入,例如大片段的插入缺失,是不推荐的。
Q2: 全外显子组测序深度的意义是什么? A: 测序深度代表了序列被探针组覆盖的次数,次数越高,测序结果的识别就越精确,后续的统计分析也就越准确。如果做肿瘤、低频突变研究,建议测序深度至少应达到150×以上。如果只看经典SNP、非低频突变,测序深度也至少应该在30×以上。
Q3: 外显子捕获效率是什么? A: 外显子测序过程中要用到杂交过程。在人的染色体上有许多与外显子有同源性的部分,这些有同源性的部分很可能在杂交过程中也被捕获下来。所以,测到的序列中,有一部分不是外显子序列。我们把测序得是外显子的部分占全部测序序列的比列称为捕获效率。捕获效率的高低不影响数据质量,只影响数据的有效比例。
|
爱泼斯坦-巴尔病毒相关性肝内胆管癌的各项特征研究
|
合作单位:中山大学肿瘤医院 发表期刊:《journal of hepatology》 影响因子IF:20.582 |
研究背景
| 爱泼斯坦巴尔病毒(EBV)是一种疱疹病毒,主要引起急性传染性单核细胞增多症。EBV影响非常广泛,全世界90%以上的人口受到了其感染,但后果却截然不同。EBV引起的疾病通常有区域差异:腺热主要发生在欧洲和北美,通常影响青少年或年轻人。在赤道非洲,伯基特淋巴瘤与EBV感染有关。在中国南部地区,以及东南亚,这种病毒经常导致鼻咽癌、鼻癌和喉癌,但也会导致其他类型的肿瘤,这篇文章主要解析EBV导致的肝内胆管癌的特征。 |
材料方法
|
303例原发型ICC肿瘤样本,进行免疫组化、免疫荧光、荧光原位杂交等指标检测。另外,从中挑选了10对癌-癌旁样本进行100X外显子测序。 文章主要从临床病理特征、肿瘤微环境以及基因组学特征对爱泼斯坦-巴尔病毒相关性肝内胆管癌(EBValCC)的各项特征进行解析。其中,基迪奥生物为该项目提供了外显子测序以及个性化生物信息分析服务(即基因组学特征分析),用高质量的生物信息和优质的售后服务,保障客户高水平文章发表。 |
研究结果
|
基因组学特征分析 文章对肿瘤中常见的三类体细胞突变进行汇总和讨论,另外也对样本中分离的EBV病毒进行了测序和分析。外显子测序会发现大量突变,但大部分突变是没有生物学意义的。对于肿瘤研究,就需要通过体细胞突变分析、驱动基因分析等,结合突变注释和基因注释,来一步步缩小目标基因的范围。 |
|
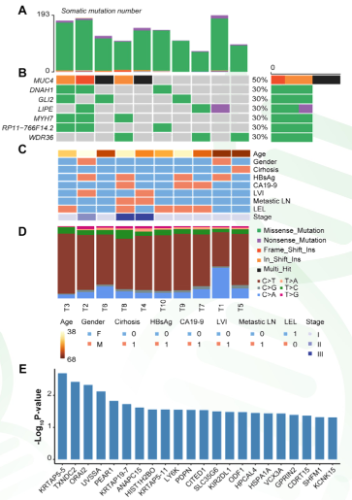
(1)体细胞突变分析(SNP/indel) 通过对10对EBVaICCs癌和癌旁样本的100X外显子测序,发现了3353个体细胞SNP和indel体细胞突变。SNP包括1346个非沉默突变和2007个沉默突变,显示出高突变率(中位数: 4.4个突变/Mb)。发现至少有三名患者的7个基因受到非沉默突变的影响,包括MUC4,DNAH1,GLI2,LIPE,MYH7,RP11-766F14.2和WDR36。主要的体细胞突变类型是C: G >T:A转换和C: G >A: T转换。此外,利用MuSigCV软件预测了EBVaICC中的22个潜在驱动基因(图1)。值得注意的是,与其他EBV导致的肿瘤相比(包括胃癌、鼻咽癌),EBVaICC显示出不同的突变模式。这说明虽然这些肿瘤都来自EBV感染,但癌基因的驱动机制并不相同(或许治疗策略也要不同)。
|
图1 EBVaICC的SNP/indel体细胞突变。(A)每个EBVaICC样本的体细胞突变基因数。(B)EBVaICC中频繁突变的基因(至少在三个样本中鉴定)。(C)根据样本列出临床病理特征。(D)每个EBVaICC样本的主要体细胞突变类型。(E)图中显示了由MuSigCV算法识别的22个潜在驱动基因,x轴显示突变类型,y轴显示估计的突变。 |
|
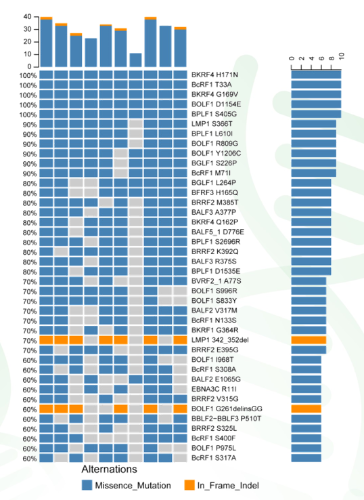
图2 所有病例EBV病毒的高频突变基因 |
(2) 体细胞突变之CNV突变分析 比较高频的拷贝数丢失包括染色体7q34 (频率30%)和14q11.2(频率50%)的丢失。没有发现染色体臂级别的拷贝数增加。缺失基因中,PRSS1缺失(三个病例中发现)被报道是胰腺炎的致病原因。对CNV相关基因的通路也进行了Kegg富集分析。 (3)体细胞突变之SV分析 通过FISH实验发现了FGFR2基因的融合,但通过novoBreak算法并没有在外显子测序数据中发现该融合突变。 (4)病毒基因组突变分析 作者也从样本中分离了EBV病毒株,并进行测序和组装。在肿瘤样本的EBV的基因组数据中频率排名前40的非同步突变见图2。所有病例的EBV病毒都在BKRF4(H171N),BcRF1(T33A),BKRF4(G169V),BOLF1(D1154E)和BPLF1(S405G)等基因存在突变。 |
|
参考文献 Huang, Yu-Hua, et al. "Clinicopathologic features, tumor immune microenvironment and genomic landscape of Epstein-Barr virus-associated intrahepatic cholangiocarcinoma." Journal of Hepatology (2020). |