|
大多数基因组中的染色质都紧紧盘绕在细胞核内,但也有一些区域经染色质重塑后呈现出松散的状态,这部分无核小体的裸露DNA区域被称为开放染色质(open chromatin),而DNA复制和基因转录都发生在这些区域。染色质的这种特性叫做染色质的可接近性(chromatin accessibility),通过研究细胞特定状态下开放的染色质区域可以在DNA水平上了解其转录调控,ATAC-seq就应用于此。
ATAC-seq(Assay for Transposase Accessible Chromatin with high-throughput sequencing,染色质易开放区域测序)利用Tn5转座酶切割染色质的开放区域,并加上测序引物进行高通量测序,通过生物信息分析鉴定转录因子结合位点和核小体区域位置,从而为研究基因调控、DNA 印记等提供有效的方法。 |
|
技术路线

分析内容
|
标准信息分析
(需提供参考基因序列、参考基因组序列及基因注释结果)
1 测序评估: 测序质量评估、碱基组成与质量分析、过滤信息统计
2 比对分析:比对基因组统计、Reads在染色体上的分布、插入片段大小分布
3 Peak分析:Peak calling、Peak长度分布、Peak深度分布、Peak富集倍数分布
4 Peak注释:Peak 相关基因分析、Peak在基因功能元件上的分布、Peak相关基因的GO/Pathway功能富集分析
5 TF-motif 分析
6 核小体定位分析
7 多样品间的差异peak分析:样品相关性分析、差异peak统计、差异peak在基因功能元件上的分布、差异peak相关基因分析、差异peak相关基因GO/KEGG功能富集分析
|
|
样品要求/项目周期
请咨询当地销售或拨打电话:020-84889324(医学)、020-84889314(农学)了解详情。 |
|
[1] Thurman R E, Rynes E, Humbert R, et al. The accessible chromatin landscape of the human genome[J]. Nature, 2012, 489(7414): 75.
[2]Wu J, Huang B, Chen H, et al. The landscape of accessible chromatin in mammalian preimplantation embryos[J]. Nature, 2016, 534(7609): 652.
[3] Sen D R, Kaminski J, Barnitz R A, et al. The epigenetic landscape of T cell exhaustion[J]. Science, 2016, 354(6316): 1165-1169.
[4] Qu K, Zaba L C, Satpathy A T, et al. Chromatin accessibility landscape of cutaneous T cell lymphoma and dynamic response to HDAC inhibitors[J]. Cancer Cell, 2017, 32(1): 27-41. e4.
[5] Corces M R, Granja J M, Shams S, et al. The chromatin accessibility landscape of primary human cancers[J]. Science, 2018, 362(6413): eaav1898.
|
|
Q1:ATAC与ChIP都可用于研究转录因子在DNA上的结合位点,这两种技术有什么差别? A:不同于ChIP技术,ATAC技术主要通过检测染色质开放区域寻找转录因子的DNA结合motif,Chip技术则是利用转录因子的特异性抗体进行免疫沉淀(IP),富集获的转录因子结合的DNA片段。ChIP技术针对单一转录因子寻找结合位点,ATAC技术则能够挖掘结合到染色质开放区的转录因子信息。
Q2:ATAC-seq 是否需要设置生物学重复? A:通常建议每个分组中设置至少3个生物学重复,以保证获得尽可能准确的结果,如生物重复间个体差异大的,建议增加生物学重复数量。
Q3:ATAC-seq的工作原理? A:核小体致密的地方,转座酶无法进入,而开放区域转座酶可以进入并切割下暴露的DNA,连接上能够被识别的测序接头的DNA片段被逐一分离出来,用于二代测序。因此,ATAC-seq可以获得全基因组范围内的正处于开放状态的染色质区域信息,基于开放区域信息进行peak扫描,分析组间差异开放性区域及差异转录因子挖掘。
|
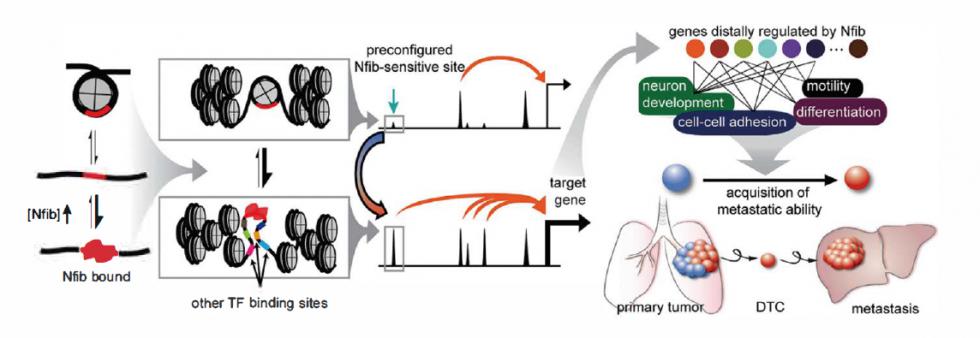
案例一: Nfib通过广泛增加染色质易接近性 (CA ) 促进小细胞肺癌的转移
|
为探究小细胞肺癌 ( S C L C ) 的转移机制,对4只模型小鼠的原发癌和转移癌 (肝)进行A T A C - seq。测序数据显示转移癌中染色质开放性普遍提高,并且差异C A 区域主要在基因远端的调控元件上,显著富集了N fi b 转录因子结合位点。结合高C A样本中N fib位点的拷贝数增加、转移癌中N fi b 的表达量显著增加、以及N fib 的C H I P - seq信号与染色质开放信号正相关的证据,锁定了关键基因N fib与S C L C 的转移密切相关。体内外的敲除与过表达功能实验证明了N fi b对于维持癌细胞的侵袭能力和克隆生长是必须的。进— 步的机制研究发现N fi b通过基因远端的调控元件增加了染色质的开放性,促进了促转移神经元基因的表达程序。 |
图I Nfib通过广泛增加染色质易接近性(CA)促进小细胞肺癌的转移 |
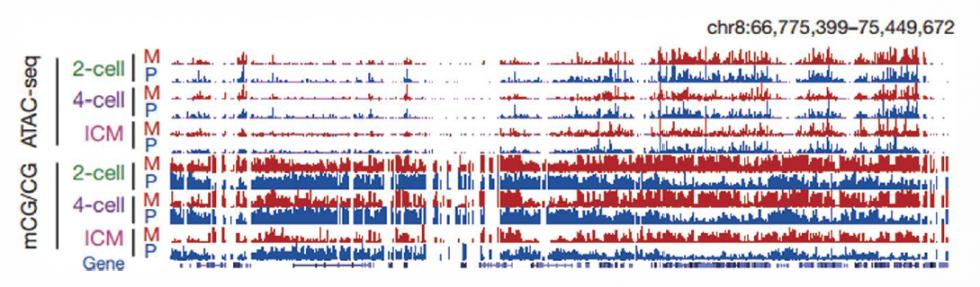
案例二:小鼠早期胚胎发育的染色质开放性图谱
|
图2 父本和母本基因组上ATAC-seq与DNA甲基化信号分布 |
为了研究小鼠胚胎植入前发育的染色质开放性变化,取 2- cell, 4-cell, 8-cell 胚胎,内细胞团(IC M ) , 胚胎干细胞 (m E S C ) 样本,进行 A T A C - seq。发现发育早期D N A 甲基化在母本基因组中普遍要高于父本,但染色质开放性在两个亲本间却相似。在胚胎发育早期,表达活跃基因的 T E S 区检测到很强的A T A C - seq 信号,可能是协助转录终止或促进转录的增强子区域。发现启动子与远端 p eak 内都富集了转座子重复序列,这些重复序列可能作为启动子或增强子参与了胚胎的基因组开放区域调控。对远端peak( 潜在enh an cer) 区域进行转录因子结合位点(m otif) 富集分析,发现m otif 富集呈现胚胎发育时期特异性。对 IC M 特异表达的— 个转录因子G A T A 4 进行R N A i 功能实验与R N A -seq , 证明了其是参与 I C M 细胞分化的关键调控因子。 |
参考文献
|
[l] Denny S , Yang D , Chuang C H , et al. Nfib Promotes Metastasis through a Widespread Increase in Chromatin Access如lity[J]. Cell, 2016, 166(2):328-342 [2] Wu J, Huang B, Chen H, et al. The landscape of accessible chromatin in mammalian preimplantation embryos[J]. Nature, 2016 |