水稻栽培种与野生种之间的基因流 返回
水稻栽培种与野生种之间的基因流

物种驯化之后,由于地理和人为选择等因素产生生殖隔离,逐渐形成栽培种、野生种等不同的种群。然而,地理上临近的群体之间仍然会发生一定程度的基因流。基因流可以使具有不同等位基因的种群产生新的遗传组合,它们可能具有更强的生存力和适应力。
基因流与种群间的地理距离之间存在很大的相关性,空间距离越短,发生基因流的概率越大,而空间距离远的种群间可能只有很小的基因流甚至没有基因流。其它种群基因的流入可以直接改变种群原有的基因频率,影响遗传结构,并使得种群间在遗传结构上趋于一致。
野生水稻(Oryza rufipogon)一直被认为是亚洲栽培稻的野生祖先,也是科学家改良水稻品种时需要的重要种质资源。科学家通过对大量野生稻和栽培稻基因组数据的深入挖掘,发现野生稻中有大量的栽培稻基因成分,甚至部分“野生稻”就是近期野化的栽培稻。
亚洲不同地区的野生稻群体,其遗传成分和本地的栽培稻成分有很大的相关性,这说明当前的野生稻通过广泛的基因流与栽培稻联系在一起,随着栽培稻共同演化。
研究背景
亚洲栽培稻是古老的广泛消费的主要粮食作物之一,它的驯化和种植促成了亚洲农业文明的兴起。9000多年前,栽培稻已从野生水稻中驯化出来,包括japonica和indica等多个品种群。
水稻亚群的起源数量一直没有达成共识,一种是单一起源模式,由于后期的驯化多样化创造了不同的亚组,另一种是多驯化起源,这些群体之所以含有共同的驯化基因是由于基因流或各自独立地选择。
这些争议存在的主要原因是栽培稻的野生祖先早已灭绝,而现存的野生稻受到栽培稻的大量基因流影响,迄今为止还没有研究过从栽培稻到野生稻的基因流数量以及这种基因流对于野生稻的影响程度。
研究材料
203 个驯化水稻品种和 435 个野生水稻品种
研究思路

研究结果
1、驯化水稻和野生水稻的群体结构
通过对203 个驯化水稻品种和 435 个野生水稻品种的全基因组数据进行群体结构分析。在野生稻种群中,确定了6个亚组(图1),分别表示为Or-A、Or-B、Or-C、Or-D、Or-E和Or-F。
四种成分(Or-A,Or-B,Or-C和Or-D)是野生稻独有的,当K从2增加到3时,Or-A组分是第一个出现在野生稻中,该组分具有广泛的地理分布。当K=5时,Or-B出现,它几乎完全在中国发现,可能代表籼稻和粳稻的野生祖先。当K = 6导致Or-C的出现,其主要发现在南亚和东南亚,Or-D几乎完全存在于印度支那半岛和孟加拉国等。
最后两个亚组(Or-E和Or-F),它们分别含有aus和indica这两个栽培种的遗传成分。

图1 驯化水稻和野生水稻的群体结构分析
2、栽培稻与野生稻之间的基因流
野生水稻中Or-E和Or-F含有相应驯化亚组(aus 和 indica)的遗传组分这个现象可以通过两种可能的假设来解释。
第一,Or-E和Or-F分别是aus和indica驯化过程中的祖先群体代表,第二,它可能是由驯化稻与相应的野生群体之间的基因流造成的。为了测试这些假设,研究者首先把所有的栽培稻和所有的野生稻进行一对一的地理距离和遗传距离之间的相关性分析,发现地理上接近的栽培稻和野生稻也倾向于具有更高的遗传相关性(图2),不仅如此,在较小的区域内(如印度和孟加拉国)也能发现这种相关性。
这种相关性可以解释为各个国家的水稻独立驯化,这显然是不可能的,因此,更有可能是这些地区的驯化稻和野生稻之间存在大量的本地基因流。

图2 栽培稻与野生稻在地理和遗传距离上的相关性
作者分析了两种已知的驯化相关基因sh4和PROG1,并在野生稻中检测它们是否出现,同时在栽培稻中检测野生等位基因是否出现。
这两个基因在之前的研究中被证明是负责从野生稻到栽培水稻的关键形态转化:sh4编码序列中的突变(G→T)导致稻谷的破碎减少,PROG1中的遗传变异使栽培稻从匍匐转变为直立。通过sh4位点的单倍型聚类分析,所有驯化的水稻品种仍然被分配到单一组分(图3),这表明它们具有高度相似的单体型。
然而,来自野生稻种群的94个样品(占所有野生样品的21.6%)也被分配到驯化组分中,说明它们在sh4中具有驯化的等位基因,PCR检测结果也证明了这一点。
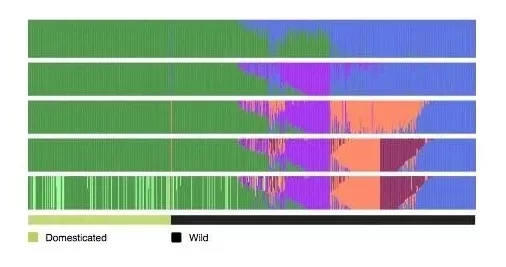
图3 sh4基因所在区域的群体结构分析
这些“野生”种质含有驯化的等位基因可以通过两个假设来解释:栽培稻的渗入或共同祖先的变异。如果是栽培稻渗入的话,这些野生稻个体在sh4基因座处应该具有驯化相关的选择性清除信号;如果这些野生种质从其祖先处获得驯化等位基因,则不应该显示驯化相关的选择性清除信号。
为了测试这个假设,作者对携带sh4驯化等位基因(以下称为WRDS)的野生稻进行了局部检测,发现核苷酸多样性减少了四倍,与栽培稻相似。此外,Tajima’s D检测也显示了近期发生的选择性清除。WRDS和驯养稻之间的遗传分歧降至0,而WRDS与野生、栽培种与野生的差异都很高(图4)。
这些结果表明,在sh4位点,WRDS和栽培稻之间的遗传相似性是由于栽培稻向野生稻转移了驯化等位基因。这种携带驯化sh4单倍型的野生稻仍然具有野生稻的种子破碎表型,说明野生稻种群发展出了一种或多种补偿机制,以应对驯化稻持续的sh4基因流。

图4 sh4单倍型在野生稻和栽培稻之间的差异
当对PROG1基因座进行分析时,发现113个野生稻种质(占所有野生稻样本的26.0%),携带驯化等位基因prog1。与Sh4观察到的模式相似,核苷酸多样性减少,Tajima's D为-2.42。
此外,其他野生稻亚群的104个种质在PROG1或sh4处携带驯化等位基因,表明基因流是稳定的,不仅仅受限于野生稻亚组的某个群体。
在野生稻种群中的sh4 / PROG1位点发现了207个驯化等位基因,而在栽培稻中很少能找到野生等位基因(n = 3),这说明基因流动是不对称的,其主导方向为从栽培种到野生种。这也与二者不同的花器官结构一致,栽培稻的花是闭合的,异花授粉困难,而野生稻具有外露柱头的花和更高的异交率。
3、栽培稻的野化及适应性选择
叶绿体基因组分析发现,大量的野生稻含有栽培稻的叶绿体(图5),说明目前的很多野生稻是由古老的栽培稻野化而来,然后在适应当地野生环境的过程中产生分化。
那么,野生稻在适应过程中有一些基因必然会受到选择因,作者用FST方法鉴定野生稻和栽培稻之间高度分化的基因。这些基因的GO分析显示,长日照光周期性相关基因是被显著富集的。HD1是基于光周期依赖性开花的主要数量性状基因座的基因,是野生水稻Or-E基因组中多样性减少最显著的区域之一。
HD1可能是水稻野化过程中受选择的目标基因,它的非同义突变有助于水稻适应野生栖息地的开花时间。这些结果进一步说明野生稻是由古老栽培稻野化而来。
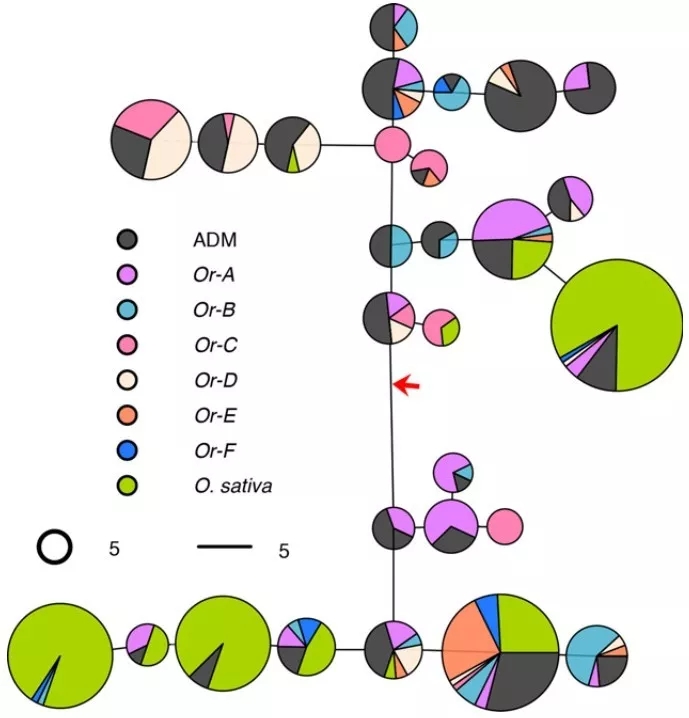
图5 野生稻和栽培稻的叶绿体单倍型分析
4、栽培稻向野生稻的基因流向
利用五个野生稻种群和四个栽培稻种群研究水稻之间的基因流。栽培稻亚群始终表现出更强的遗传漂变,可能是因为它们在驯化和人工选择过程中经历了巨大的瓶颈。
只允许一个迁移事件(m = 1),观察到indic到Indochina野生稻种群(ICW)的混合,贡献了Indochina野生稻46%的DNA。当m = 2时,观察到从Indochina野生稻到china野生稻的大量基因流,这可能是原始野生稻和栽培稻混合为china野生稻的结果。
当m = 3时,china野生稻与Indochina野生稻、Archipelago野生稻聚为一类,但得到来自japonica祖先的大量基因流(49%)(图6),还有从aus到TRJ的19%的admixture事件,表明这两个亚群之间具有大量相似的遗传血统。可能的是,中国发现的Or-B成分不是“真正的”野生成分,而是野稻和古代粳稻的混合物。

图6 亚洲驯化水稻的基因流
总 结
该研究通过分析203个栽培稻和435个野生稻来研究野生稻的遗传血统,发现多数现代野生稻通过基因流与栽培稻发生大量混杂,甚至可以推断目前的野生稻只是代表不同阶段野化的栽培稻。
与这一假设相符的是,很多野生稻在栽培稻的驯化基因位点表现出选择性清除痕迹,而在开花基因位点又表现出近期的选择性适应证据,这些都可能与栽培稻的野化有关。该研究提出野生水稻群体是一个杂交群,它们通过持续的、广泛的基因流与栽培稻相连接。
参考文献
Wang H, Vieira F G, Crawford J E, et al. Asian wild rice is ahybrid swarm with extensive gene flow and feralization from domesticatedrice[J]. Genome Research, 2017.
更多的动态,可继续关注我们网站,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























