KEGG命名系统的介绍 返回
KEGG数据库的内容很多,核心是KEGG pathway。数据均来自文献,通过手绘路径图集。
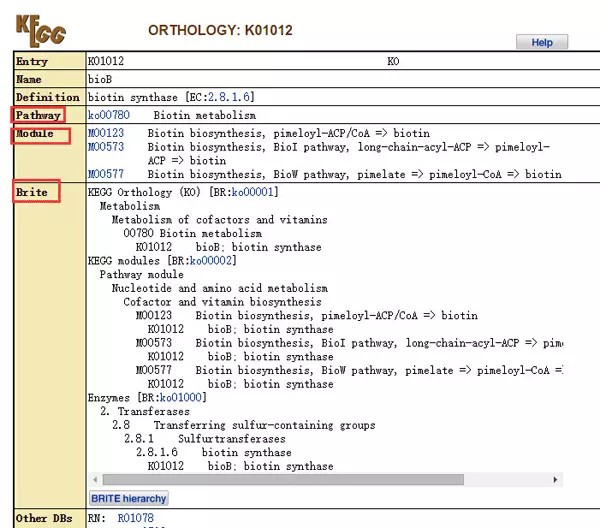
如上图,KEGG除了核心内容pathway,还有对数据库的信息分类(如,Brite),或pathway的组成元件的信息(Module、orthology)。这些信息往往会交叉出现。
下面,我们以“生物素合成酶”为例:
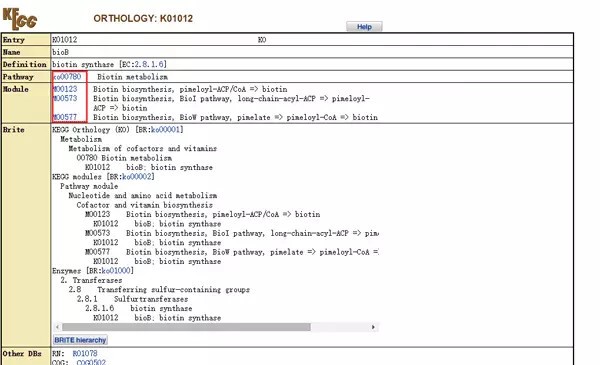
从上面的图可以注意到这些蓝色小字,为什么有些是K开头的,有些是KO开头的,还有些是M或者C开头的,我们来解释一下:
1、K+num(基因ID号,表示在所有同源物种中具有相似结构或功能的一类同源蛋白)。
如:K01012=>生物素合成酶(备注:K建议大写)
2、ko+num(代谢通路名称,表示一个特定的生物路径)
如:ko0078 => 生物素代谢通路 (备注:ko小写)
3、M + num(模块名称)
M00123 => 生物素合成模块
4、C+num(化合物名)
如C00120:生物
5、E -.-.-.-(酶名)
EC2.1.1.116 => 生物素合成酶(其实也就是k01012)
6、R+num(反应名)
7、RC+num(反应类型)RP+num(反应物质对)
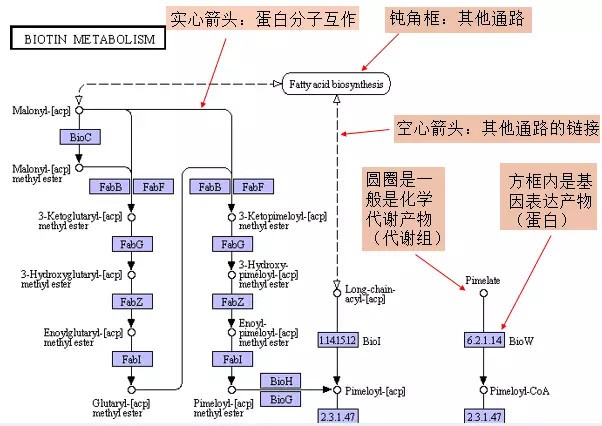
生物素代谢通路中的符号
KEGG Modules:功能单元

m开头。这些模块往往是pathway的重要组成部分(但也只是一部分)。包括:路径模块、结构复合物、功能集、信号模块等。
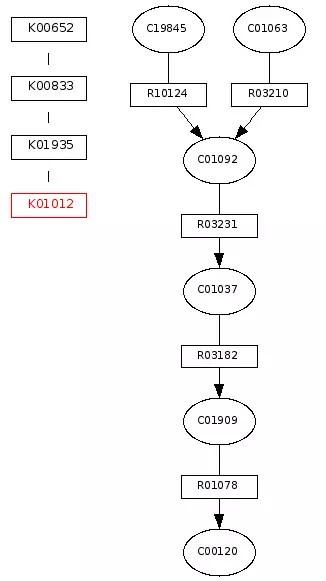
这里的模块展示的就是生物合成的过程(上),相当于这个pathway的局部地图(简化版)。
KEGG BRITE
KEGG中,信息的层级分类系统,KEGG会从不同的角度分层级。如下图,从pathway,modues和Enzynes三个角度,分析了这个基因的从属关系。

从酶的角度考虑,这个酶属于转移酶的大类,更次级的分类是巯基转移酶….这样层层分类,来展现这个酶的信息。
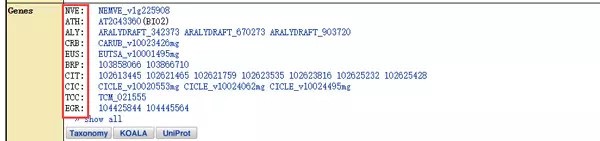
另外,界面还包括了这个基因在不同物种中的信息,前三个字母是这个物种拉丁名的简写,例如ATH代表拟南芥。
更多基迪奥精彩原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!


























