应用领域
技术路线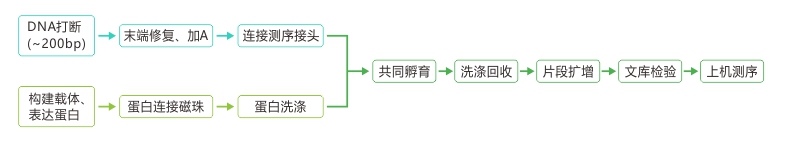
分析内容
基迪奥执行的部分转录因子
| 转录因子家族 | 物种 | 转录组因子家族 | 物种 | 转录组因子家族 | 物种 |
| GATA | 水稻 | FT | 水稻 | COL | 矮牵牛,黄瓜 |
| ARF | 油菜 | SMAD | 斑马鱼 | HB | 拟南齐,桃子 |
| ARF17 | 玉米 | TCP7 | 苹果叶片 | MYC | 丹参,橡胶树 |
| B3 | 蓖麻 | MBF2 | 小菜蛾 | HSF | 玉米,木薯 |
| BES | 地钱 | RAV | 陆地棉 | LBD | 杨树,桃子,红豆杉 |
| LEC1 | 油菜 | RWP-RK | 珍珠粟 | GRAS | 小麦袧橘,菜心 |
| GT | 草莓 | FT | 水稻 | TALE | 大豆,玉米,番茄 |
| IRP | 刺参 | PRRs | 大豆 | TCP | 大豆,木著,水稻,菊花 |
| SBP | 水稻 | TT1 | 油菜 | MADS | 水稻,玉米,甘薯,番茄 |
| CAMTA2 | 番茄 | Tox3 | 小鼠 | SPL | 相橘,揪树,玉米,甜橙,薄壳山核桃 |
| CCT | 薏苡 | TAGATA | 小麦 | HD-ZIP | 樱桃,玉米,板栗,番茄,称猴桃 |
| DBB | 水稻 | WUS | 菊花 | MYB | 文冠果,番木瓜,酵母,陆地棉,马铃薯 |
| PHD6 | 大豆 | YABBY | 冬瓜 | NAC | 梅花,水稻,玉米,甘薯,白菜,康乃馨,捞猴桃,梨 |
| PR | 高粱 | ZFP4 | 生菜 | C2H2 | 玉米,番茄,真菌,荔枝,慧叹,坛紫菜,半滑舌鳎 |
| ERK2 | 牡蛎 | CNC | 长牡蛎 | ERF | 大豆,小麦,桃子,番茄,茶树,疲萝,龙眼,矮牵牛无花果,平邑白茶叶片 |
| RUNT | 马氏珠母贝 | AP2 | 花生,蓖麻 | bHLH | 玫瑰,虾夷扇贝,大豆,木薯,杨树,水稻,茶树,火龙果,矮牵牛,马铃薯 |
| STE | 家蚕微孢子虫 | APRR2 | 油菜,番茄 | bZIP | 辣椒,白杨,半滑舌鳎,草苺,水稻,菊花,谷子,坛紫菜,矮牵牛,文冠果,酵母 |
| NF-kB | 太平洋牡蛎 | BZR | 小麦,文冠果 | WRKY | 木瓜,桃子,油菜,玉米,甘庶,甜橙,番茄,白梨,芒果,苹果,水稻,番木瓜 |
| RWP-RK | 珍珠粟 | DOF | 菊花,丹参 | MBD | 涡虫,丹参,木棉,相橘,梨花,油松,油菜,甘荒,甜橙,番茄,盐齐,节瓜,茶叶,草苺,黄岑 |
参考文献



























