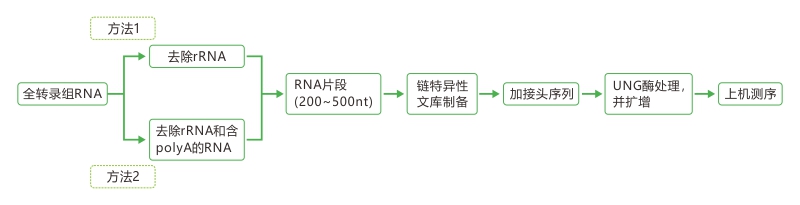
技术路线
分析内容
|
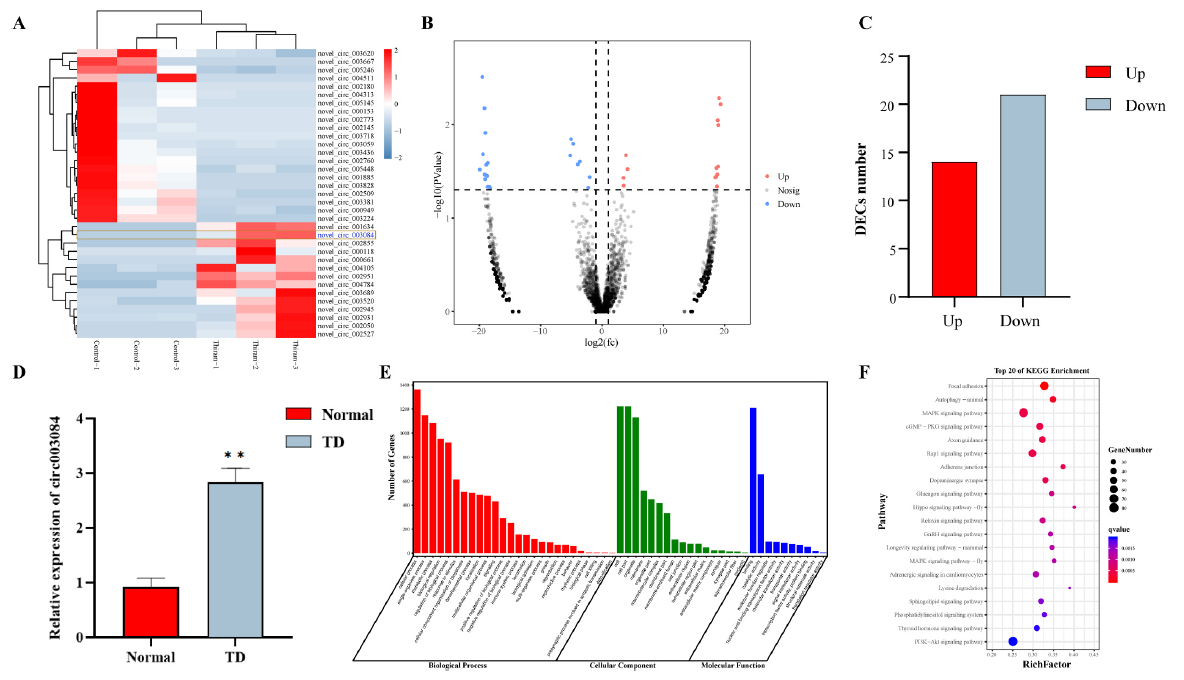
1. 标准信息分析
a) 测序数据评估;数据比对统计,测序饱和度分析,测序随机性的统计分析,Reads在参考基因上的分布分析
b) 已知 lncRNA 的识别与注释
c) 转录本重构与新lncRNA预测
d) LncRNA 表达量的统计
e) LncRNA 样本关系分析
f) LncRNA差异分析
g) LncRNA 家族分析
|
2. 高级信息分析
a) lncRNA 位置分类(Intergentic lncRNA,Intronic lncRNA,Cis-antisense lncRNA)
b) 候选lncRNA的保守性分析
c) 贯穿分析(需同一样品的不同水平数据,如跟sRNA测序结果关联。可参考miRNA-TF-gene network分析方案, 或者miRNA-LncRNA关联分析)
d) 候选基因分析(某种表达模式基因或者非编码基因,或者转录因子,需指定)
e) 定制图表(可按老师提供的参考文献或者要求制作)
f) 定制个性化分析(老师提供方案或者参考文献)
|
全转录组研究Thiram暴露对鸡软骨细胞增殖与分化的调控机制