分析内容
|
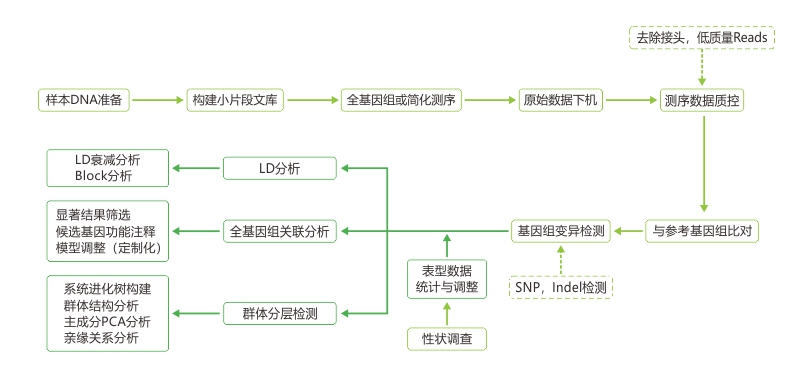
1.标准分析 1.1原始数据过滤 1.2数据比对 1.3SNP与indel检测 2.高级分析 2.1连锁不平衡分析 2.2群体结构分析(PCA、进化树、structure分析) 2.3多态性分析 2.4候选基因提取与突变注释 |
3.个性分析 3.1表型分析 3.2基因型分析 3.3GWAS分析(简单广义线性模型分析、广义线性模型分析、混合线性模型分析、K+Q线性模型分析)
|
GWAS鉴定玉米籽粒镉积累的候选基因
|
全基因组关联分析(Genome-wide association study,简称GWAS)是一种在群体水平研究表型-基因型关系的研究策略。GWAS是在特定群体中检测全基因组水平数以百万计的分子标记(例如SNP标记、CNV标记)基因型信息的基础上,开展群体中个体表型与基因型的相关性分析,从而解析影响复杂性状的基因变异。随着测序价格的不断下降,全基因组重测序结合GWAS分析,快速定位控制复杂性状的功能性遗传突变,解析对应性状的遗传调控机制,已经称为一种主流的研究方式。 |
|
1. 动植物各类数量性状的研究;
2. 人类各类表型/复杂疾病研究。
|

分析内容
|
1.标准分析 1.1原始数据过滤 1.2数据比对 1.3SNP与indel检测 2.高级分析 2.1连锁不平衡分析 2.2群体结构分析(PCA、进化树、structure分析) 2.3多态性分析 2.4候选基因提取与突变注释 |
3.个性分析 3.1表型分析 3.2基因型分析 3.3GWAS分析(简单广义线性模型分析、广义线性模型分析、混合线性模型分析、K+Q线性模型分析)
|
|
样品要求/项目周期
请咨询当地销售或拨打电话:020-84889324、020-84889314了解详情。 |
|
参考文献 [1] Lin H, Wang F, Rosato AJ, Farrer LA, Henderson DC, Zhang H. Prefrontal cortex eQTLs/mQTLs enriched in genetic variants associated with alcohol use disorder and other diseases [published online ahead of print, 2020 Jun 4]. Epigenomics. 2020;10.2217/epi-2019-0270. [2] Loh NY, Minchin JEN, Pinnick KE, et al. RSPO3 impacts body fat distribution and regulates adipose cell biology in vitro. Nat Commun. 2020;11(1):2797. Published 2020 Jun 3. [3] Maroteau C, Kalhan Siddiqui M, Veluchamy A, et al. Exome sequencing reveals common and rare variants in F5 associated with ACE inhibitor and ARB induced angioedema [published online ahead of print, 2020 Jun 4]. Clin Pharmacol Ther. 2020;10.1002/cpt.1927. doi:10.1002/cpt.1927. [4] Howell AE, Robinson JW, Wootton RE, et al. Testing for causality between systematically identified risk factors and glioma: a Mendelian randomization study. BMC Cancer. 2020;20(1):508. Published 2020 Jun 3. doi:10.1186/s12885-020-06967-2
|
|
Q1: GWAS的材料最少要多少份? A:不能一概而论。因为GWAS是统计学结果,哪怕目前只有50株材料,最后能得到一个显著位点的话,结论依然是有效的。理论上群体越大,越有能力检测到微效的QTL。因为GWAS分析的目标往往是数量性状,如果材料少于二三百株的话,可能无法检测到中低效应的QTL。
Q2: GWAS能关联多个性状,一般能关注多少个性状? A:对于性状数量是没有限制。
Q3: 单样本测序深度与群体大小的关系,哪个对GWAS分析结果的影响更大? A:单样本测序深度低,主要会导致基因型缺失,从而影响性状定位,而基因型缺失可通过缺失填充来弥补。而样本量少,检测到显著关联的SNP位点几率会小很多,相对风险较大。综合考虑,同样成本条件下做选择,建议增加测序样本量,适当降低单样本的测序深度。
|
GWAS鉴定玉米籽粒镉积累的候选基因
|
合作单位:中国科学院植物研究所 发表期刊:Journal of Hazardous Materials |
|
目的:许多重质膳食的生产和使用几乎不可避免地将镉(Cd)释放到环境中,产生Cd污染物,对食品和人体健康产生不利影响。开发一种有效的粮食作物镉含量评价方法,可为其毒性预测和污染控制提供有效途径。本研究从全基因组水平探索玉米籽粒Cd积累的基因型-表型关系,并利用机器学习和线性统计方法构建全基因组关联研究(GWAS)辅助的基因组预测模型。
取材:219个不同的玉米自交系群体
结果:(1)使用rrBLUP的GP模型表现优于Bayes A和RF,具有更高的GP精度(rMG)和更低的平均绝对误差值。 (2)定位的SNP注释基因,做GO富集分析发现,主要富集在转运蛋白活性、生物过程调节。 (3)4个基因(GRMZM2G064563、GRMZM2G455491、GRMZM2G340578和GRMZM2G153488)与水稻Cd积累相关基因OsCd1、OsHMA3、OsCCX2和CAL1具有较高的序列同源性。 (4)鉴定了重金属相关结构域蛋白(HMA)、天然抗性相关巨噬细胞蛋白(NRAMP)、atp结合盒转运蛋白(ABC)、主要促进因子超家族(MFS)、钠钙交换蛋白、黄条样蛋白(YSL)、金属硫蛋白样蛋白、防御素蛋白、锌转运蛋白、铜转运蛋白、空泡铁转运蛋白等46个基因。 (5)将GP与GWAS相结合可作为准确评价Cd浓度的有效策略,为加快低Cd粮食作物的选种周期和解决环境Cd污染问题提供有益的指导。
图1.GP的SNP标记
参考文献: Yan H, Guo H, Xu W, et al. GWAS-assisted genomic prediction of cadmium accumulation in maize kernel with machine learning and linear statistical methods[J]. Journal of Hazardous Materials, 2023, 441: 129929. |









