|
10x Genomics单细胞转录组测序(single cell RNA-seq, scRNA-seq),是在单个细胞水平对 mRNA 进行高通量测序的一项新技术,其能够以高通量和单分子分辨率研究单个细胞表达谱,揭示复杂细胞群体的异质性,避免单个细胞的基因表达信号被群体的平均化所掩盖。
10x Genomics系统具有高通量、周期短、适用范围广等优势,一个样品可进行1000~10000个细胞转录组分析;6.5min可完成上万细胞的封装,一天内完成单细胞捕获、反转录扩增与建库;相较其他平台,单个细胞的测序成本低;适用于绝大部分的细胞类型,包括生殖细胞、胚胎细胞、血细胞、神经细胞、肿瘤细胞、免疫细胞、干细胞等。
|
|
1. 发育生物学:胚胎、神经、脑等各种组织;
2. 肿瘤异质性与耐药性研究;
3. 干细胞分化潜能研究;
4. 免疫方向;
5. 细胞图谱绘制。
|

分析内容
|
标准信息分析
1.测序数据统计与评估:各个样本测序数据基本质控(reads数、测序饱和度等)、各个样本数据比对(细胞数目统计、reads基因组比对率等)、基因表达定量;
2.单细胞亚群分类与分类结果可视化:细胞过滤、细胞亚群分类、分类结果可视化(TSNE图);
3.亚群上调表达基因分析:上调表达基因筛选、上调基因表达分布、上调基因蛋白质互作网络分析、上调表达基因基因GO/KEGG功能富集分析、DO基因疾病富集分析(人)、Reactome通路富集分析(人、大鼠、小鼠);
4.基因集变异分析GSVA分析;
5.膜蛋白注释(物种必须具备蛋白序列库)。
|
|
高级信息分析 1. 拟时分析(monocle):推断出发育过程细胞的分化轨迹或细胞亚型的演化过程; 2. 拟时分析(PAGA):计算细胞亚群之间的连通性,检验后构建PAGA网络图,筛选差异表达基因; 3. 细胞通讯分析(物种限制:人、小鼠和大鼠):通过统计不同细胞类型中受体和配体的表达及配对情况,推断不同细胞之间的相互作用; 4. 转录因子分析(物种限制:人、小鼠和果蝇):体现不同细胞/细胞类型之间的转录因子调控活性差异,绘制转录调控网络图 5. 细胞周期分析(物种限制:人、小鼠和大鼠):推断单个细胞的细胞周期; 6. 单细胞CNV分析(限肿瘤):通过基因的相对表达量来推测单细胞染色体区域的CNV;肿瘤克隆亚型分析、CNV分布分析; 7. RNA速率分析:通过mRNA生成和降解过程的动态变化,推断细胞分化发育轨迹。
|
|
Omicsmart定制化 在线报告分析 1. 细胞过滤:样本细胞数目抽平、低质量细胞过滤、细胞质控总览; 2. 细胞集筛选:流程细胞集筛选、目标细胞集筛选; 3. 基因集筛选:新建基因集、合并基因集、基因筛选; 4. 细胞亚群分类分析:分类堆叠图、分类tSNE图、分类UMAP图; 5. 细胞注释:在线完成40+物种、200+组织类型、4000+细胞类型的自动注释; 6. 亚群marker基因分析:tSNE图(表达量和分类)、小提琴图、热图; 7. 亚群上调基因分析:tSNE图(表达量和分类)、小提琴图、热图,GO/KEGG富集分析; 8. 组间差异分析:差异基因分布、差异基因富集; 9. 细胞亚群再分群分析:子亚群分类分析、子亚群上调基因分析; 10. 拟时分析:细胞轨迹图、拟时间轴差异基因分析、分化状态差异基因分析、分化命运差异基因分析; 11. 目标基因分析:韦恩分析、富集分析。
|
|
样品要求/项目周期
请咨询当地销售或拨打电话:020-84889324、020-84889314了解详情。 |
|
参考文献
[1] Maaten L V D, Hinton G. Visualizing Data using t-SNE[J]. Journal of Machine Learning Research, 2008, 9(2605):2579-2605. [2] Adamson B, Norman T M, Jost M, et al. A Multiplexed Single-Cell CRISPR Screening Platform Enables Systematic Dissection of the Unfolded Protein Response[J]. Cell, 2016, 167(7):1867. [3] Zheng G X, Terry J M, Belgrader P, et al. Massively parallel digital transcriptional profiling of single cells[J]. Nature Communications, 2017, 8:14049. [4] Mcdavid A, Finak G, Chattopadyay P K, et al. Data exploration, quality control and testing in single-cell qPCR-based gene expression experiments[J]. Bioinformatics, 2013, 29(4):461-467. [5] Cell ranger :http://support.10xgenomics.com/single-cell/software/overview/welcome. [6] Dimitri Meistermann, Alexandre Bruneau, Sophie Loubersac, et al. Integrated pseudotime analysis of human pre-implantation embryo single-cell transcriptomes reveals the dynamics of lineage specification[J]. Cell Stem Cell, 2021, 28(9):1625-1640.e6. [7] Wolf, F.A., Hamey, F.K., Plass, M. and et al. PAGA: graph abstraction reconciles clustering with trajectory inference through a topology preserving map of single cells[J]. Genome Biol, 2019, 20(59). [8] Alessia Centonze, Shuheng Lin, Elisavet Tika, et al. Heterotypic cell–cell communication regulates glandular stem cell multipotency[J]. Nature, 2020, 584:608–613. |
|
Q:单细胞转录组、空间转录组和 bulk 转录组区别? A:三者都属于检测 mRNA 表达水平的转录组技术。区别主要在于分辨率和分析维度的不同。 普通转录组获得的是混合细胞的基因平均表达水平;单细胞转录组通过对单个细胞人为标记,获得群体内单个细胞的基因表达谱,有助于发现新细胞、挖掘细胞的异质性;而空间转录组则是直接在组织原位上进行 mRNA 捕获,同时获取细胞的空间位置信息和基因表达数据,从而进一步推动对组织原位细胞真实基因表达的研究。
Q:10X单细胞标记的细胞数目范围一般是多少?细胞标记上的数目是越多越好吗?还有捕获率偏低是什么原因? A:标记效率官方是 65%,如果悬液质量比较好,活性高,杂质少,基本都可以达到这个效率,甚至比这个效率还高。 但实际上除了跟效率有关,还跟细胞数目计数准确性有关,因为细胞计数的时候,我们数的视野是很有限的,所以,细胞计数准确性不一定都能保证,所以也会造成标记效率的波动。标记上的细胞多,那么拿到的细胞肯定也多,但是也会有另一种问题,就是单个细胞的数据量少了,所以这里要做一定平衡。捕获率低跟悬液质量关系比较大,杂质,碎片这些往往捕获率低。
Q:单细胞测序需要做重复吗?如果做重复的话,是几个重复? A:重复这问题可以参考普通转录组的演化历史。 首先一个基本逻辑,任何生物学实验都需要做重复的,这是第一点逻辑;第二审稿人也会考虑可执行性。以普通转录组为例,在 2010 年时,普通转录组非常昂贵,一个转录组好几万。那么审稿人也非常理解这一点,所以如果你不做重复的话,通常审稿人也不会太多质疑这一点;随着价格下降以后,审稿人默认大家能够承担这个实验成本了,一般就会要求做重复, 这是普通转录组的演化。即随着成本的不断下降,做重复慢慢的会作为要求多起来。 再说回单细胞,目前单细胞转录组发文章,一般不要求做重复,主要原因还是太贵。但从未来来说,可能随着价格下降,慢慢审稿人可能会要求做重复。 从单细胞测序做重复的必要性来说 ,还需要具体问题具体分析。如果只是关注基因表达, 做细胞的鉴定,然后挖掘一些目标 marker,然后做下游的验证。因为这个研究下的文章重点,其实还是需要有下游验证的。这种情况下不做重复,也没问题。 但如果文章很多结论都来自单细胞测序,那做重复会更可靠。比如关注的是细胞频率,即细胞的比例的变化。因为本身单细胞悬液制备过程中前处理、消化、制悬液以及细胞核的提取这中间其实是有误差的。如果不做重复,然后文章结论又说处理组相比对照组某个细胞比例下降了。其实这结论可能是成疑问了。当然如果单细胞测序没做重复,但后面用流式的方法,做细胞比例验证,那也可以的。 总结来说,做不做重复,还是要考虑成本以及文章的主要结论。推荐最好做 3 个。但考虑性价比的话,两个重复是性价比较高的策略。用最小的成本解决重复的问题。
Q:单细胞核测序优势 A:和单细胞测序相比,单细胞核测序的优势在于: 1)细胞核的大小远远小于原生质体细胞,这就避免因为原生质体悬液中有较大的细胞而放弃上机标记。 2)同时,细胞核提取的偏好性小于原生质体制备,能更好地代表整个组织中,真实的细胞类型组成。 3)而且单核提取过程避免了长时间对组织进行酶解消化,进而避免了对基因表达产生影响,可以获得更真实的基因表达情况。 4)有些研究可能需要跨越一个部位的不同生长发育时期,或者实验处理后不同时间点取样,整个周期可能较长,同一批次进行原生质体制备难度比较大,而细胞核制备的话,则可以把不同时期的样本,分别收集进行液氮速冻保存起来,之后同一批次进行单细胞核的制备和标记测序。 5)另外,如果使用细胞核进行单细胞测序研究,则可以应用转录组测序+ATAC 测序多组学联合标记,对每个核同时进行转录本信息以及染色质开放性信息捕获。 6)提取的细胞核,可以使用流式细胞分选等手段,去除大部分碎片杂质和次生代谢产物,获得高纯度的细胞核。 同时也要注意,对细胞核进行测序,细胞质中转录本信息就丢失了,获得的是核内转录本信息,核内的 mRNA 主要是正在加工的新合成 mRNA,并且其含量是少于胞质 mRNA 的。不过众多文献报道核 mRNA 和细胞总 mRNA 的相关性是很高的,分群效果是一致的。
Q:marker 基因怎么获得? A:1)cellmarker:http://biocc.hrbmu.edu.cn/CellMarker/,包括人的 158 个组织 (亚组织)的 467 个细胞类型的 13,605 个 Marker 基因,和鼠的 81 个组织 (亚组织)的 389 个细胞类型的 9, 148 个 Marker 基因; 2)文献梳理; 3)亚群上调表达基因推测; 4)近缘物种的同源基因; 5)基迪奥公司积累数据库
|
10X单细胞测序研究儿童结肠炎及炎症性肠病发病机制及治疗方法
|
合作单位:广州市妇女儿童医疗中心 发表期刊:《cell》 影响因子:36.216 |
| 儿童期发病的结肠炎和炎症性结肠炎(PIBD)的发病率逐年增加,该类疾病反复发作、治疗无效率高且费用昂贵。而且PIBD临床表现复杂及致病机制不清严重限制了治疗方法的选择和个性化诊疗的开展。为此,研究团队利用单细胞转录组、单细胞免疫组库和全基因组关联分析(GWAS)探究PIBD的发病机制及可用药物。 |
研究思路
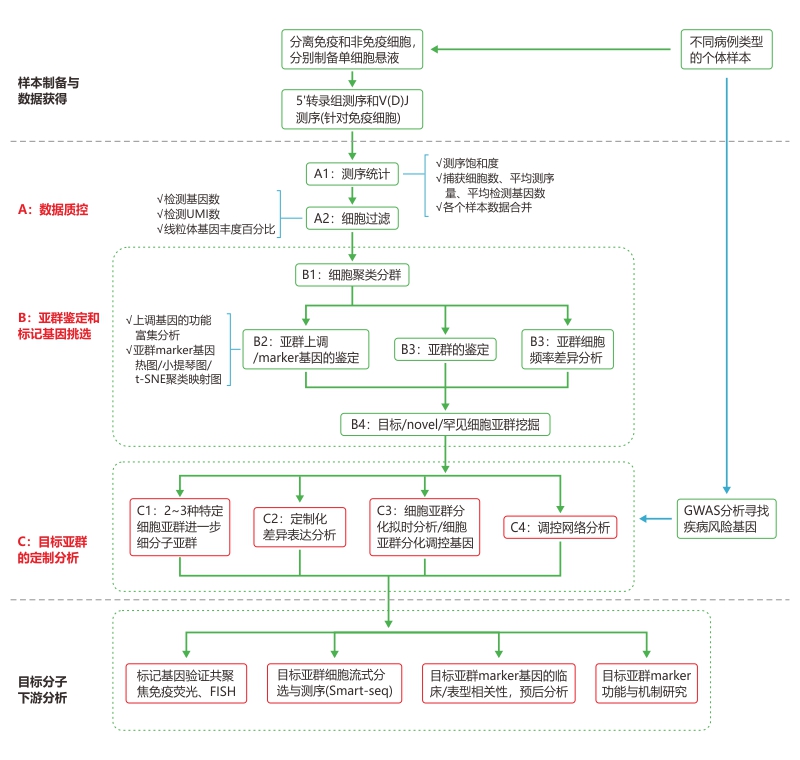
| 文章研究思路 |
研究结果
1. 队列特征与单细胞转录组图谱(分群)
|
(1)样本选择和队列分析
为了阐述不同亚型病例的致病机制并寻找治疗靶点,研究团队对17名患儿(对包括对照组在内的四组取样,如下图A)的结肠黏膜进行取样测序。研究采用FACS方法分离免疫细胞和非免疫细胞,然后进行单细胞转录组及TCR+BCR测序(针对CD45+免疫细胞同时开展转录组和免疫组库测序)。
单细胞测序结果将免疫细胞和非免疫细胞的数据合并分群,分群如下图(B)。从图中我们可以看出即包含了常见免疫细胞(包括:B细胞、T细胞、NK细胞、髓系细胞等),又包含了非免疫细胞(包括成纤维细胞、上皮细胞、内皮细胞)。
在文章的后续部分,作者将对这些亚群中的上皮细胞、成纤维细胞、髓系细胞以及T/B细胞子亚群进一步进行分析讨论。
|
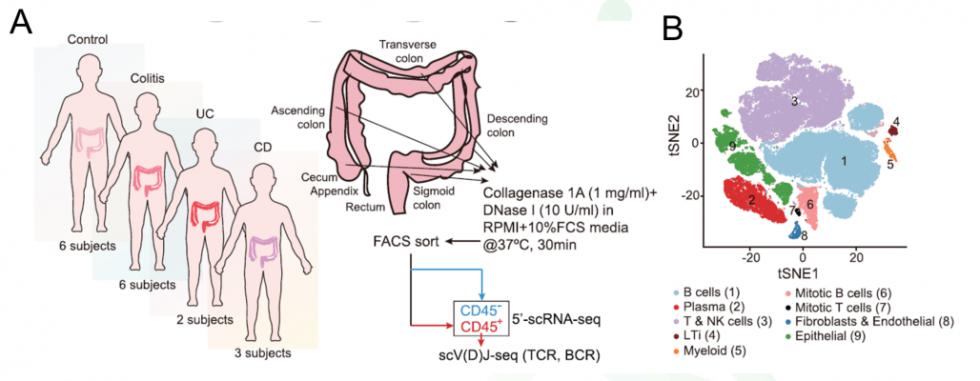
图1 队列特征与结肠黏膜单细胞图谱
|
(2)全基因组关联分析(GWAS)
为了寻找PIBD的风险基因,作者基于210个病例和614个对照进行了全基因组关联分析,定位了244个与PIBD风险基因。这部分风险基因将与后续单细胞亚群特异表达基因进行比较,分析风险基因主要在哪类细胞中起作用。
|
|
2. 上皮细胞子亚群分析:PIBD风险基因在哪些子亚群特异表达
将上皮细胞分为了10个子亚群(图2A),值得关注的是其中有两个子亚群没有被鉴定。结合单细胞子亚群分析结果,作者还讨论了PIBD风险基因(来源GWAS结果)在各个子亚群细胞以及分化轨迹中的表达情况(图2D、F)。比如CASP7特异表达与结肠上皮细胞(colonocytes),已报道可能引起肠上皮(coloncytes)凋亡;PIEZO1特异表达在Goblet细胞上,与激活的离子通道相关。 通过这一步简单的分析,就实现了传统GWAS分析与单细胞转录组数据的关联。 |
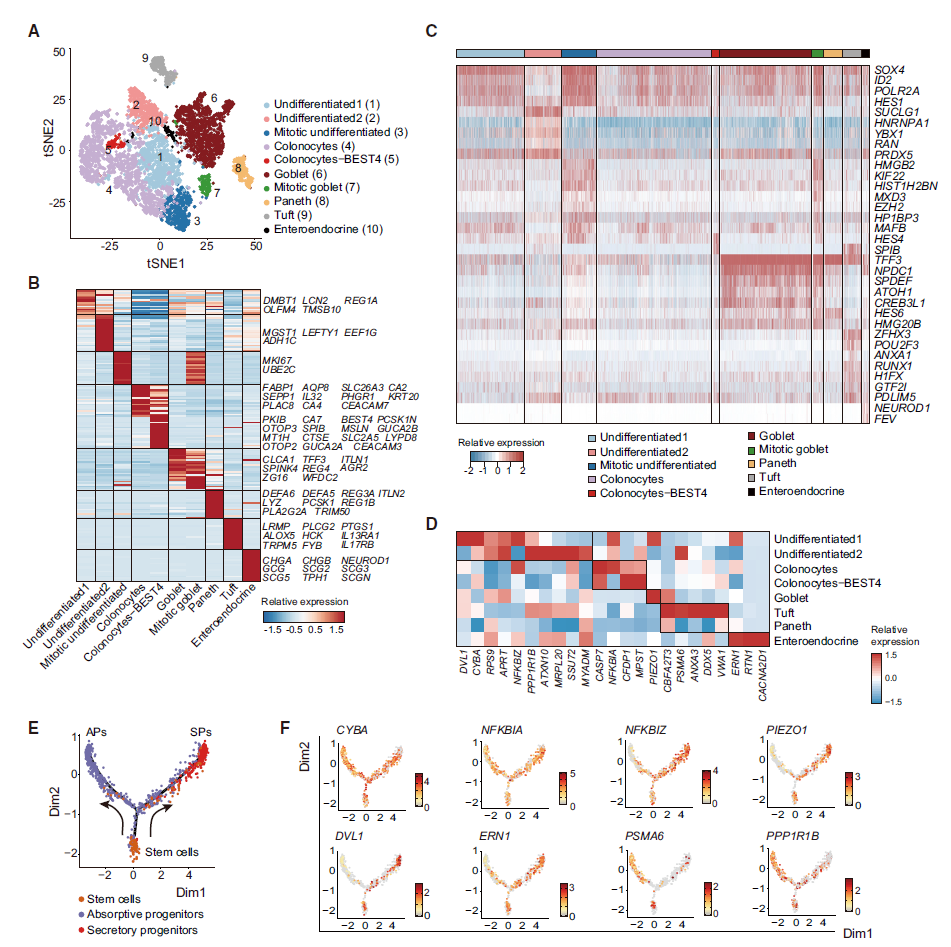
|
3. 成纤维细胞以及皮细胞子亚群分析——儿童结肠炎及炎症性肠病结肠黏膜富集炎性成纤维细胞且血管生成增加
分析得到结肠黏膜成纤维细胞共7个亚群以及和血管内皮细胞1个子亚群(图3A),分析得到8个子亚群特异表达的基因(图3B)。这些子亚群在4组样本里明显比例不同 (图3C)。值得注意的,作者不仅仅是根据前人报道的marker,也会根据子亚群特异表达的转录因子,潜在功能等信息来定义子亚群。 |
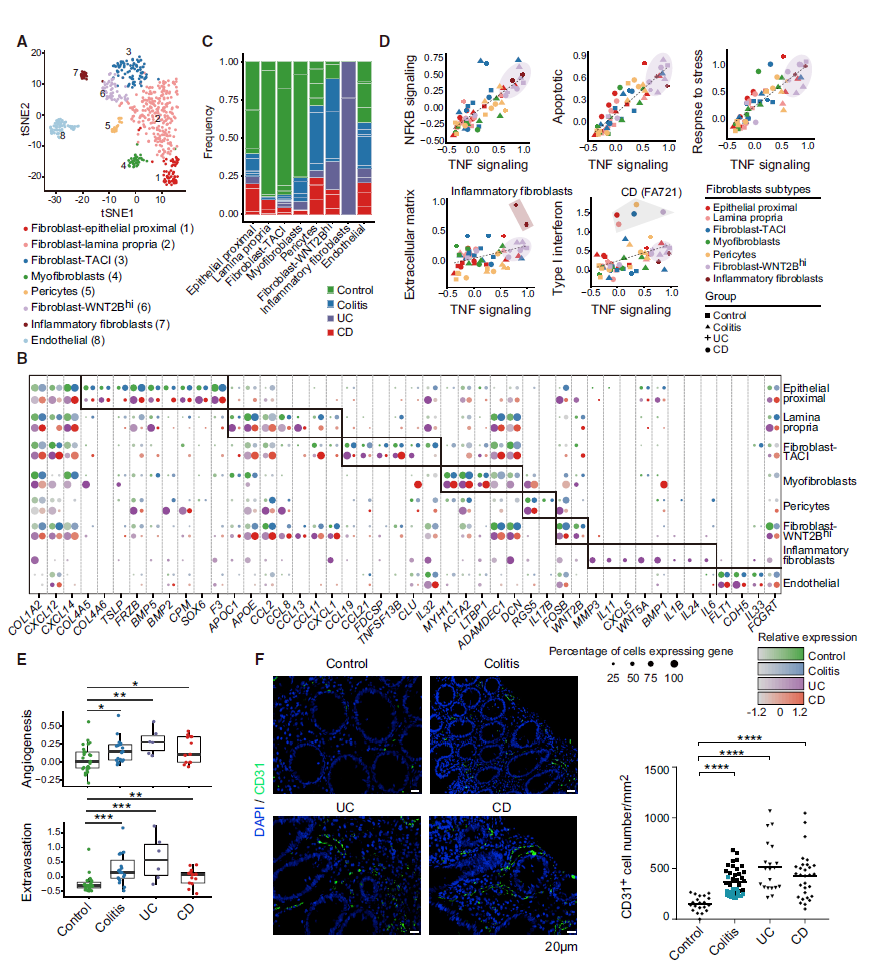
图3 PIBD与对照组的成纤维与内皮细胞组成与功能差异
|
4. 髓系细胞子亚群分析——儿童结肠炎及炎症性肠病结肠黏膜富集高炎性髓系细胞 髓系细胞在患者组中的比例显著要高于对照组(图4A)。髓系细胞全部可表达CD68,可以分为10个子亚群。通过亚群特异表达基因的pathway富集分析,作者定位了几个值得关注的核心通路(图4E),例如NF-kappa B signaling pathway, TNF signaling pathway等。 |
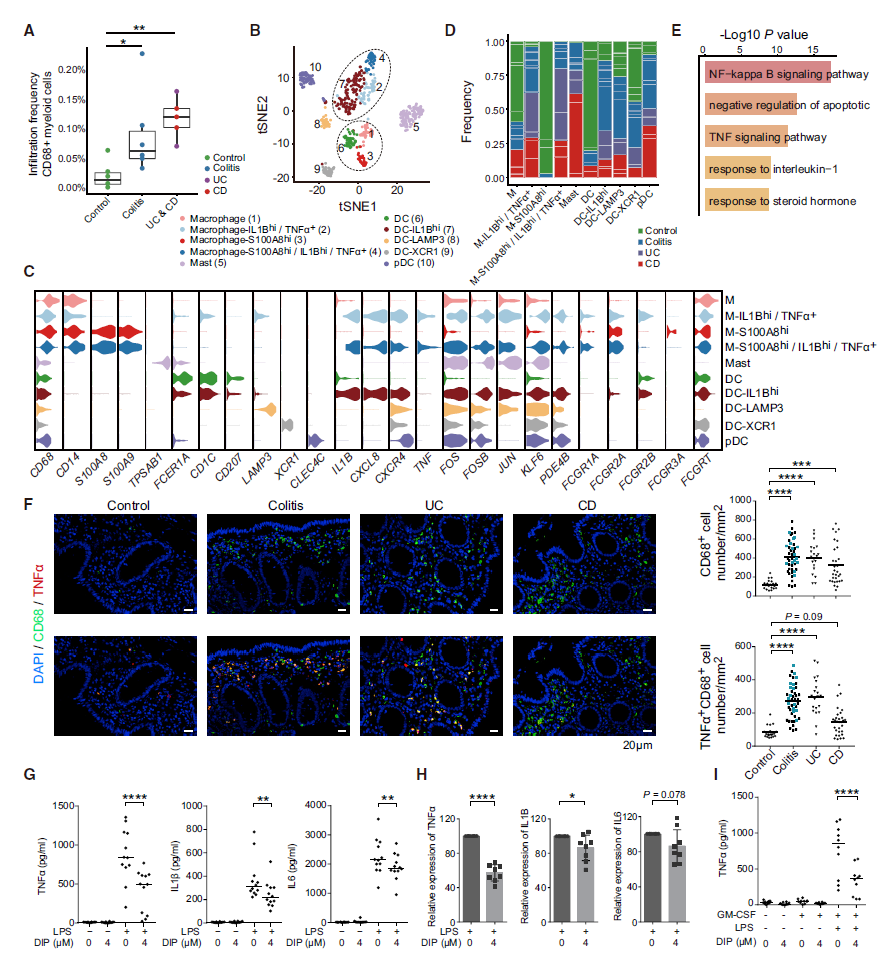
|
5. B细胞的子亚群分析——单细胞转录组以及BCR免疫组库分析
作者对两类B细胞分别开展讨论和分析:组织驻留的记忆B细胞(可以转化为浆细胞)以及浆细胞(即效应B细胞)。相对对照组,作者发现CD19+ 记忆B细胞在结肠炎组比例显著上升,分化后的B细胞——CD138+浆细胞则在IBD组显著上升(图5A)。通过子亚群分类,CD19+ B细胞分为了7个子亚群,CD138+浆细胞分为了2个子亚群(图5B),同样也分析讨论了这些子亚群的关键标记基因(图5C)。对于B细胞,该研究同时进行了单细胞BCR免疫组测序。 分析发现,在Brm-CD27lo, plasmablast(浆母细胞)和plasma cells(浆细胞)中BCR的克隆显著扩增(具有更多的BCR种类)。通过分析记忆B细胞和浆细胞共有的BCR种类所占比例,推测浆细胞的转化来源。分析发现大部分浆细胞是由Brm-CD27lo类型的记忆B细胞转化而来的(图5E)。在PIBD患者(UC和CD)组,lgG+ 浆细胞比例显著上升,这与之前报道lgG+ 浆细胞会促进PIBD病程发展一致(图5F)。最后,作者重点关注了浆细胞中在病例组高表达的与NF-kB激活相关的基因(图5G-H)。 |
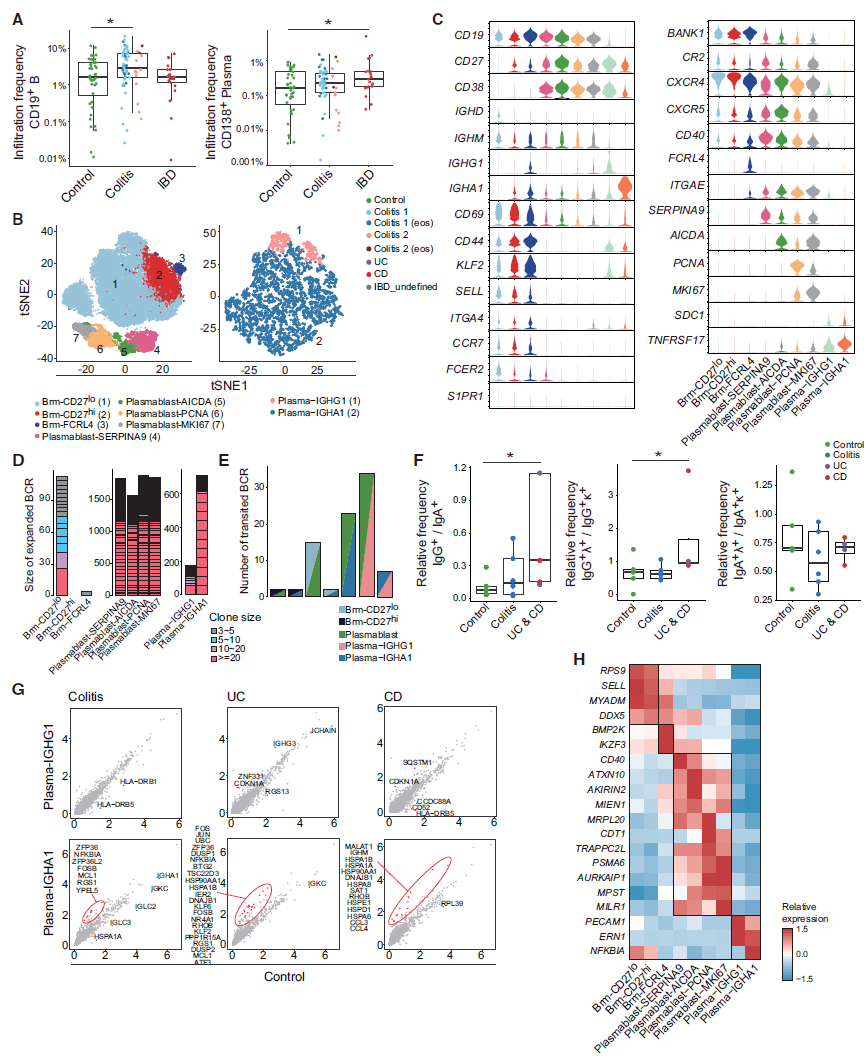
图5 B细胞子亚群转录组以及BCR分析
|
6.T细胞、NK细胞的子亚群分析——单细胞转录组以及TCR免疫组库分析 对于T细胞和NK细胞,一共又可以分为16个子亚群(图6A),作者同样整理了子亚群特异表达的基因(图6B),尤其是子亚群特异表达的转录因子(图6C-D)。类似B细胞分析,作者也分析了两大类T细胞(CD4+和CD8+)中TCR的多样性(图6E),以及通过分析不同类型T细胞共有的TCR,推测它们的转化关系(图6F)。 值得关注的是,细胞表面表达的核酸水解酶CD39 (由ENTPD1编码) 在CD8+和Vδ1+T细胞的表达在PIBD各亚型中显著降低(图6G)。为了寻找与ENTPD1相关的基因,作者采用共表达分析的方法,发现GPR65, CD247 (编码TCR zeta 链),和MAP3K8 三个关键基因与ENTPD1表达正相关。 |
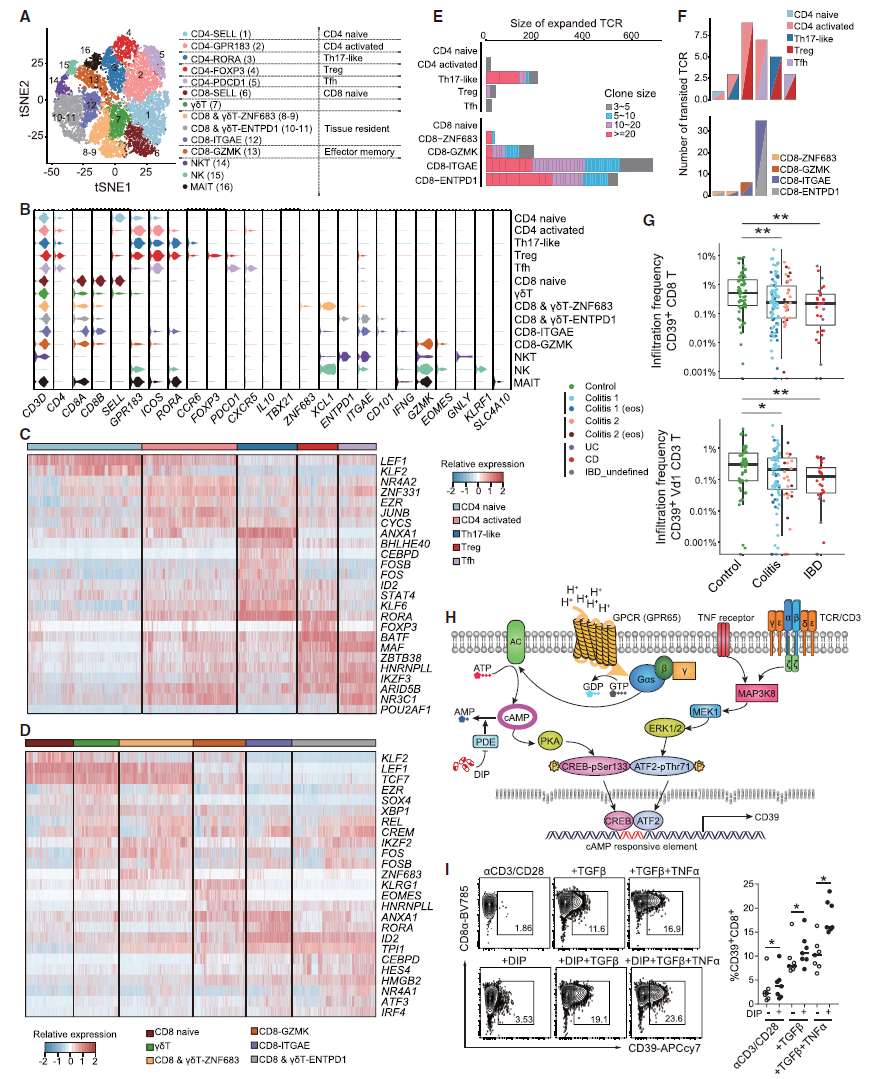
图6 T细胞/NK细胞转录组以及TCR分析
|
7. 双嘧达莫在防治儿童结肠炎及炎症性肠病方面的应用前景
研究团队利用腹腔注射双嘧达莫的方式对硫酸葡聚糖(DSS)诱导的急性肠炎小鼠进行了治疗,发现双嘧达莫可以显著改善小鼠的体重、结肠长度和结肠的组织病理结构,抑制小鼠结肠黏膜的血小板聚集和TNFα的释放(图7A-D)。
对9例患儿(8例结肠炎和1例未定型PIBD)开展了双嘧达莫的小样本临床试验,发现患者的血小板计数,结肠镜评分和临床综合评分均有明显改善(图7E)。其可能的机制是双嘧达莫通过cAMP-PKA-ERK-ATF2/CREB途径提高CD39的表达,从而抑制血小板的聚集和活化(图7F-G);同时,该药可以抑制CD68+髓系细胞及TNFα的释放(图7H),最终缓解临床症状。
|
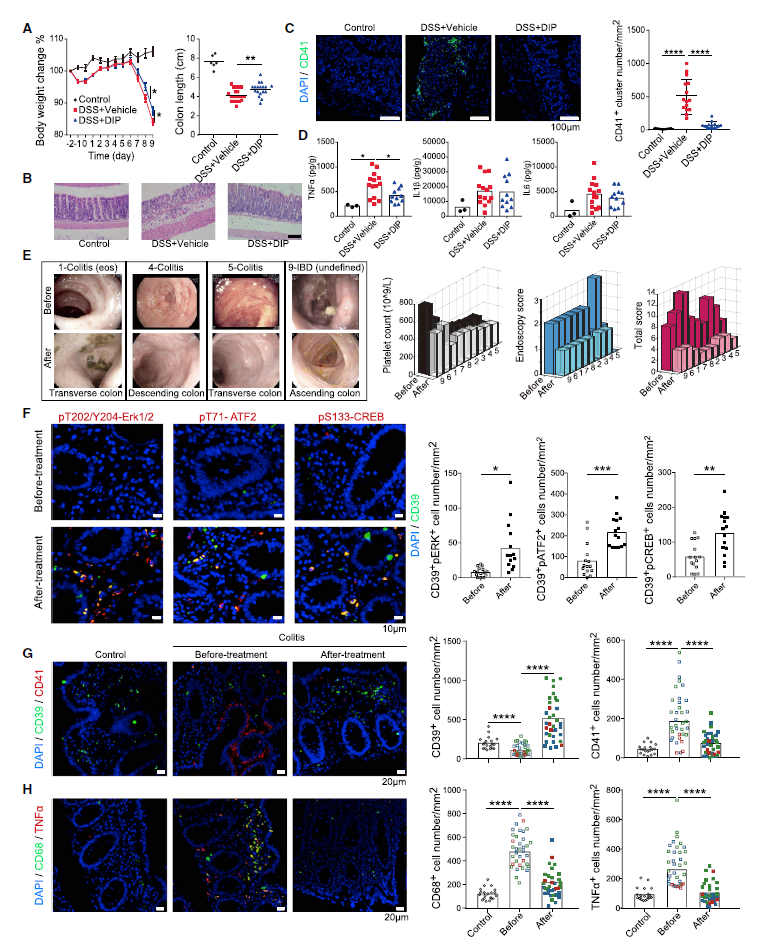
图7 双嘧达莫在临床上可以缓解结肠炎
|
参考文献 [1]Mucosal Profiling of Pediatric-Onset Colitis and IBD Reveals Common Pathogenics and Therapeutic Pathways[J]. Cell,2019,179(5). |


























