详解BinGO的用法 返回
首先我们来看看BinGO的功能
1)实现基因的富集分析
2)将富集分析的功能分类(term),以网络图的方式呈现(本质是气泡网络图)。
使用BinGO前,我们需要在在cytoscape的主界面Apps那一栏安装BinGO,在此我们就不讲解了。
一、操作
使用BinGO的操作主要有以下几步:
1)确定待富集的基因集;
2)基因注释文件;
3)GO功能分类间的层级关系文件;
4)参数设置;
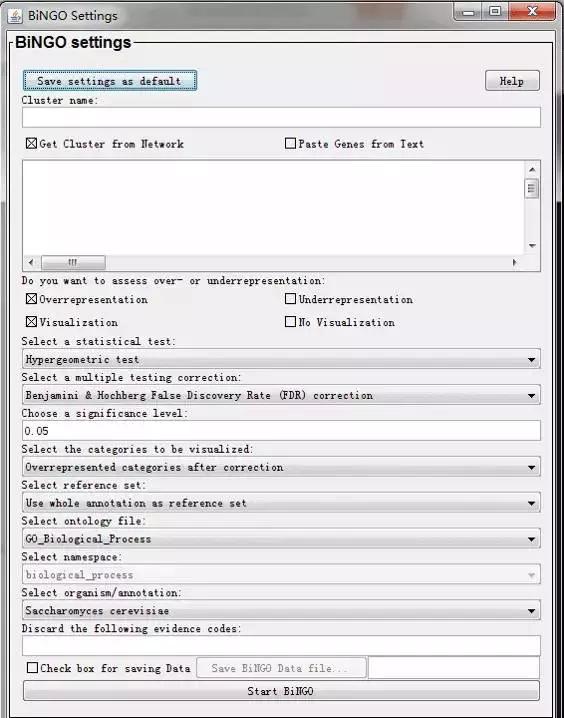
我们以模式生物拟南芥为例:
1)设置输入的基因集
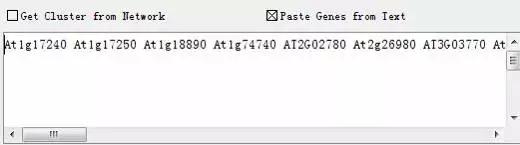
点选:Paste Genes from Test
然后将待富集的基因的名称复制到内容框中(用空格或换行符分开)。
这里使用拟南芥的10个基因来测试(10个蛋白激酶)
At1g17240 At1g17250 At1g18890 At1g74740 AT2G02780 At2g26980 AT3G03770 At3g45640 AT4G33950 At5g01810 AT5g14210 AT5g63410
2)设置基因注释
拟南芥GO注释可以从GO数据库下载。
http://geneontology.org/page/downloads
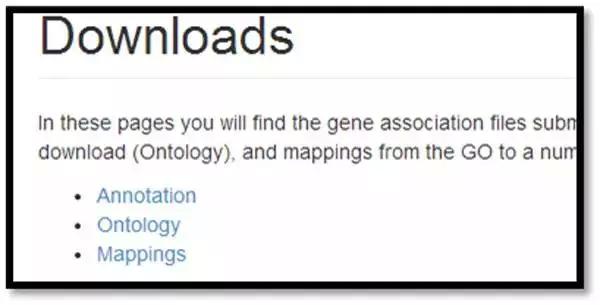
点击“Annotation”,然后从页面中找到拟南芥的基因注释,并下载
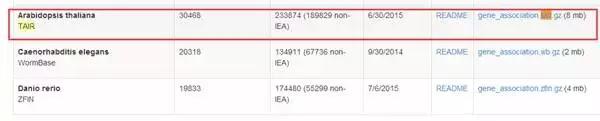
注释文件较大,但可以用excel 打开查看,这个注释文件包含的两列对这里的分析是有用的,分别是Gene ID和GO 功能注释。但文件还包含了其他信息,如symbol,基因的物理坐标信息,UniProt ID等。
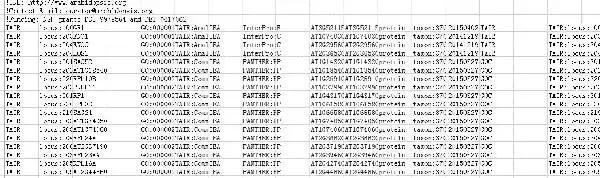
将刚刚下载的gene_association.tair 导入到 BinGO中,找到binGO的“Select organism/annotation”选框,选择“Custom”,然后选择路径导入文件“gene_association.tair”

3)设置GO功能分类关系文件
http://geneontology.org/page/downloads
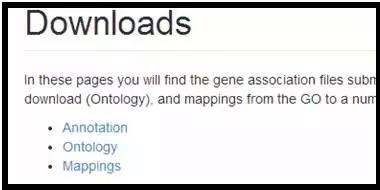
相同界面,点选Ontology,在界面内,找到“go-basic.obo”并下载。
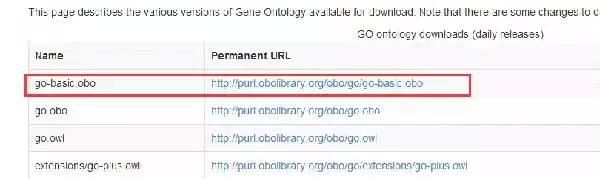
“go-basic.obo”可以使用文本文件打开。(里面记录了GO term 间的关系,依据这些关系,GO term最终将被化成网络图的形式。)
例如下图,“mitochondrial genome maintenance” is_a “mitochondrion organization”(前者是后者的某一细类)

“go-basic.obo”导入:找到 select ontology file的窗口,选择custom,然后导入。在select namespace:选择 goslim_plant(拟南芥是植物)
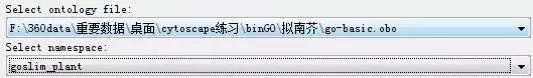
4)参数设置
选择输出文件的名称,这里设置为了my_test1;
点选“Check box for saving Data”,然后设定输出文件的路径。
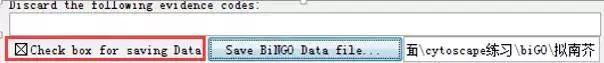
选择要输出用于画图的Go term的P值阈值(默认是0.05)

其他选择默认参数,然后点击“Start BinGO”运行。
二、结果
两个关键文件:
1)GO富集分析结果
一个文件属于弹窗,另一个存储在输出文件夹中(内容相同)。
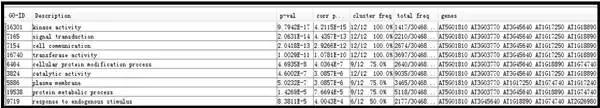
GO富集分析结果的格式
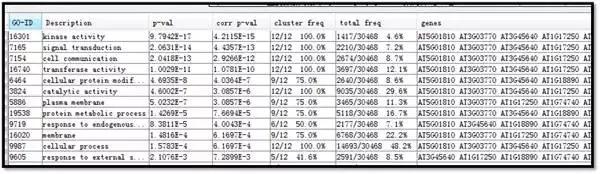
P值请参考 “Corr p-val”。第五列和第六列列出了这类功能基因在目标基因集合和全基因组基因中的比例。
另一个文本文件的内容也是相似的。
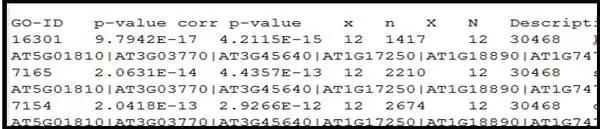
x:目标基因中此类基因的个数;
X:目标基因的总个数;
n:基因组中此类基因的个数;
N:基因组基因的总个数
2)富集到的功能分类的层级网络图
富集到的GO term按照层级关系列出网络,并按照富集程度(p值)上色。


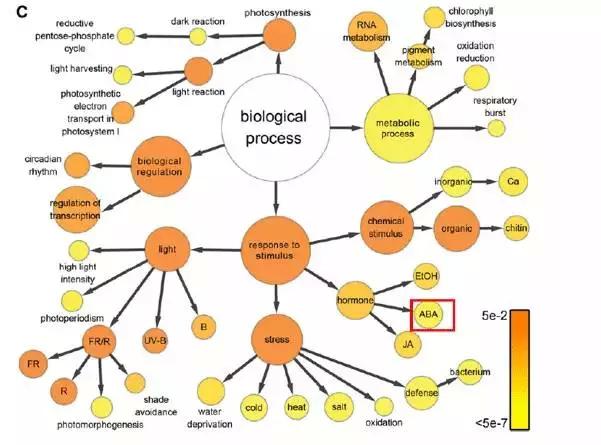
一些文章中的图片,都是用这样的方法画的。(本质是气泡网络图)
三、最后
BinGO主要针对一些模式生物,因为有现成的注释文件,如果想要用BinGO来分析一些自己研究的物种,需要手工(编程)生成注释文件,类似前文中的“gene_association.tair”。然后其他操作是相同的。
大家可以按照上面的步骤实操一下,使用中有问题,可以在基迪奥信息交流群里交流哦,群号:67185986。
更多基迪奥精彩原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























