【原创】PNAS文献 | 高粱群体遗传结构GWAS分析解读 返回
今天基迪奥跟大家解读一篇更恢弘的高粱重测序文章,样品数更多(971株),分析内容更多(群体结构、遗传多样性、GWAS定位候选基因),这篇文章是由美国康奈尔大学、国际半干旱热带作物研究所、伊利诺伊大学、美国农业部等机构的科研大牛共同完成的,发表在2013年1月的PNAS上。

首先看看文章的总体框架:
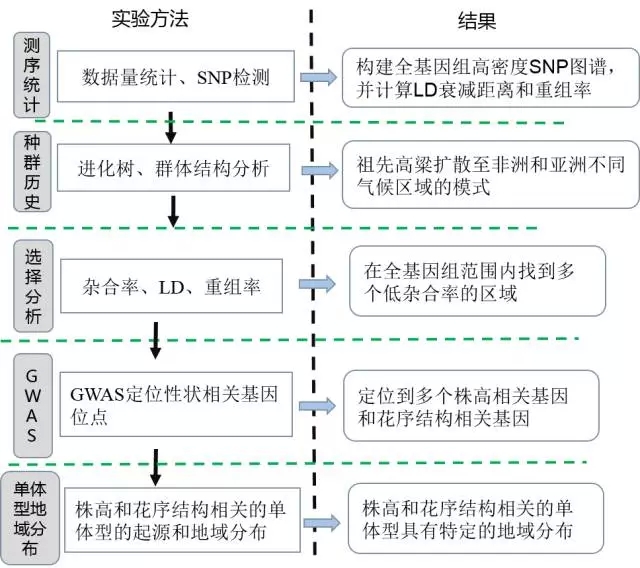
文章从不同层次、利用不同方法描绘了高粱基因组的遗传多样性。
接下来,解析文章最重要的三部分结果。
1. 种群历史和群体结构分析
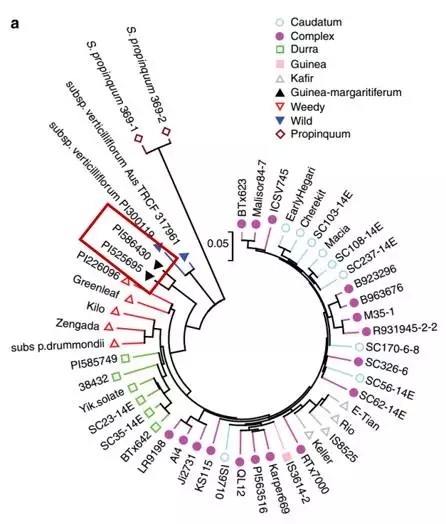
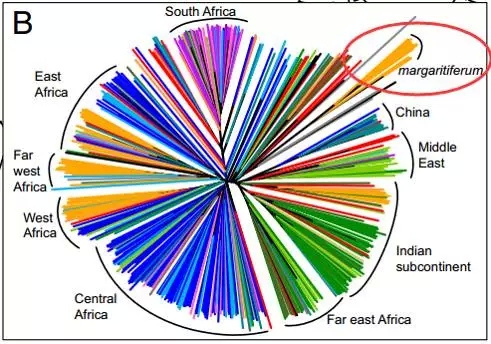
实验材料的971株高粱是来自世界各地不同地域的,要研究高粱的遗传多样性,首先要搞清楚高粱的群体结构。因此,对这971株高粱进行了进化树分析和群体结构分析,结果显示高粱的群体结构与高粱形态和地域起源都有关,揭示了祖先高粱扩散至非洲和亚洲不同气候区域的模式。

图1 高粱种质起源和遗传关系
2. 选择压力分析
为了研究高粱驯化与分化的基因选择信号,对全基因组进行了核苷酸多样性检测(π, θ)、Tajima’D检验、杂合率(He)计算。
结果:
全基因组扫描显示很多区域呈现低杂合率(Mb级别的),表明高粱基因组的很多区域潜在受到了进化选择。在之前鉴定到的6个与淀粉代谢相关的驯化候选基因中,两个位于这些低杂合率区域,分别是bt2和o2(图2 A)。
在携带早熟和矮小等位基因的高粱导入系(conversion lines)中,也同样观察到几个杂合率降低的区域,这些区域与已知的株高相关基因(Dw2, Dw3, Dw1)和成熟相关基因(Ma1)共定位(图2 B)。
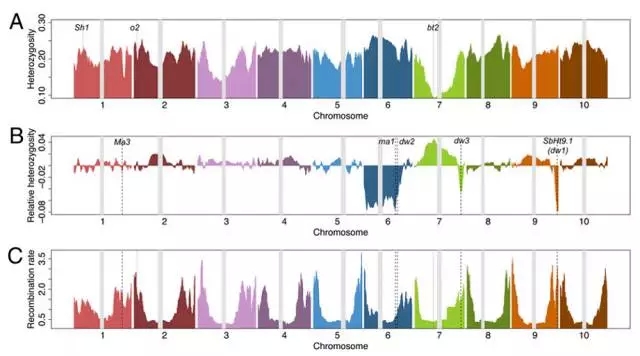
图2. 全基因组SNP变异图谱
3. GWAS分析筛选农业性状相关基因
然后作者利用全基因组关联分析(GWAS)的方法筛选与株高和花序结构相关的基因。
结果:
(1) 定位到三个株高相关已知基因:dw1, dw2, dw3(图3)
(2) 定位到多个花序结构相关基因,这些基因与玉米、水稻、拟南芥的花调控因子同源,包括GDD1,APO1等(图4)
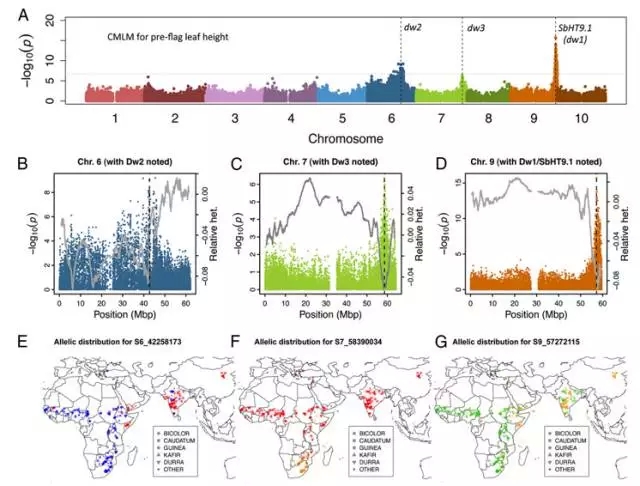
图3. 株高相关性状GWAS关联分析
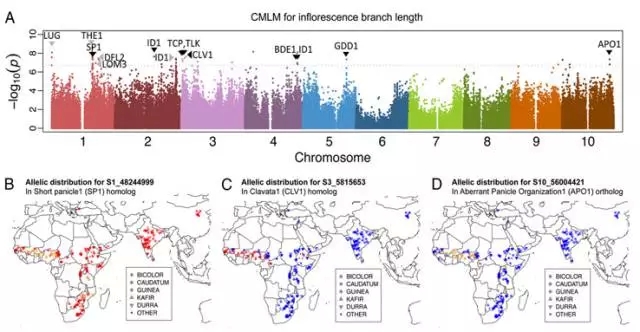
图4. 花序相关性状GWAS关联分析
注意:文章只用336株来自美国的高粱群体(SAP)进行GWAS分析,是为了减少群体结构对分析结果的干扰。
总 结
从研究单位、样品数和样品来源就知道这篇文章是一项花了重本的大范围合作科学研究,虽然没有对候选基因进行功能验证,但由于这项研究采用了不同的方法、从不同层面揭示了来自世界各地的高粱的群体结构及遗传多样性、定位了很多农业性状相关基因,为高粱的分子标记辅助育种改良提供了非常宝贵的资源,因此文章发了9.8分的PNAS了。
上周基迪奥跟大家分享了一篇发表在nature communications(NC) 上"高粱重测序选择压力分析"的文章 ,下面我们来比较这篇PNAS高粱群体分析文章和上周分享的NC高粱选择分析文章的异同点。
相同点:
GBS和全基因组重测序(不同的方法)可以得到相似的结论:两篇文章都对不同品种高粱的群体结构进行了分析,都得出西非品种Guinea-margaritiferums经历了独立的驯化事件的结论。
NC文章
PNAS文章
不同之处:
(1)研究精度不同
这篇文章使用的是GBS的测序策略,由于GBS只对酶切位点附近进行测序,大概只能测到3%的基因组,因此基因组上不在酶切位点附近的很多SNP位点都检测不到,使得不管是选择压力分析还是GWAS筛选性状相关基因,都只能定位到区间,区间内的序列就无法得知了。而NC文章采用全基因组重测序的方法,能够得到完整的基因一致性序列,因此能够在不同种群间比较这些基因的变异情况、并且进行进化分析。
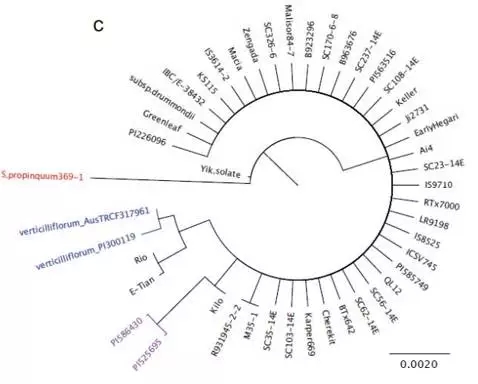
NC文章驯化候选基因dep1进化树
(2)思路不同(这是最关键的问题)
今天解读的文章比上次解读的NC文章早发了7个月,而且对高粱的种群历史群体结构、选择进化分析、GWAS分析筛选候选基因等工作都完成了,而且涉及的样品数高达900多株。
如果某个物种研究领域的大牛,进行了一次如此广度的筛查,是不是意味着其他同行就无法超越了呢?当然不是!NC的高粱选择压力分析文章就给了我们一个很好的范例。
PNAS完成如此全而广的工作后,后续的文章如果再和它拼材料的多样性或者分析条目的多样性就是死路一条。拼“全”是不可能了,所以NC文章的核心点在于设计的精巧和对细节讨论的细致性。
一般来说,大部分驯化种的驯化过程都有两步:
第一步:野生种(wild)→地方种(landrace)
第二步:地方种(landrace)→良种(elite)
很显然,第一步一般是早期人类完成的,而第二步是近现代或现代人类进一步选育完成的。这两个步骤的育种目标和育种强度是不同的,对应受选驯化选择的基因也不同。NC文章设计的巧妙之处是按照这两个步骤选取了3类材料(野生种、地方种、改良自交系),然后讨论这两个步骤中哪些基因受到了驯化选择。最后效果不错,至少NC的影响因子还是高于PNAS嘛。
与这篇PNAS文章的比较,NC文章的设计思路可能更值得大家借鉴——从细节着手深入讨论一个问题。
最后,建议做群体遗传方面研究的老师仔细研读这两篇文献,能学到这类研究一般的方法和思路。
更多基迪奥的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























