小众混池测序基因定位方法MutMap-Gap 返回
MutMap-Gap是什么?
之前的MutMap和MutMap+方法,我们都需要知道亲本的参考序列。但是在大多数情况下,研究的品系可能与数据库中该物种的参考基因组有一些区别。比如这个岛国团队,他们研究的水稻品种是“Hitomebore”,但是水稻的参考基因组来自另外一个品种“Nipponbare”。
由于计算SNP-index时需要比对到亲本的参考序列上,如果EMS诱导的突变发生在一个参考基因组没有的基因上,那么用上述的MutMap和MutMap+就无法找出该候选基因。而MutMap-Gap的方法就是结合了MutMap和de novo组装的一种基因定位方法,从而解决参考基因缺失位点上的基因突变问题。

MutMap-Gap的方法究竟是什么?下面我们详细介绍。
MutMap-Gap的原理
首先我们要将所研究品系的野生型和参考基因组比对,得到所研究品系的参考基因组。然后利用EMS诱变,再进行MutMap分析,得到SNP-index图。对位于SNP-index图peak区域内的基因进行分析,如果在这个区域内找不到跟突变性状相关的基因,那么突变位点很有可能就在品系特有基因区域内(图A)。
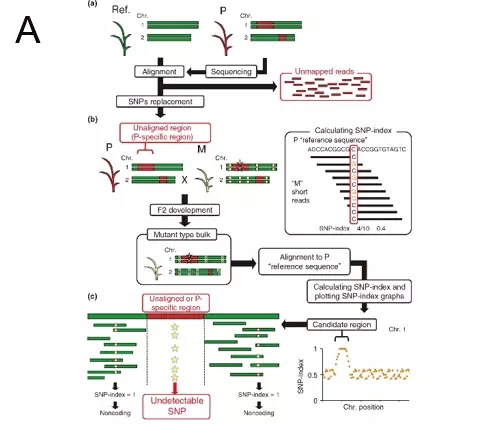
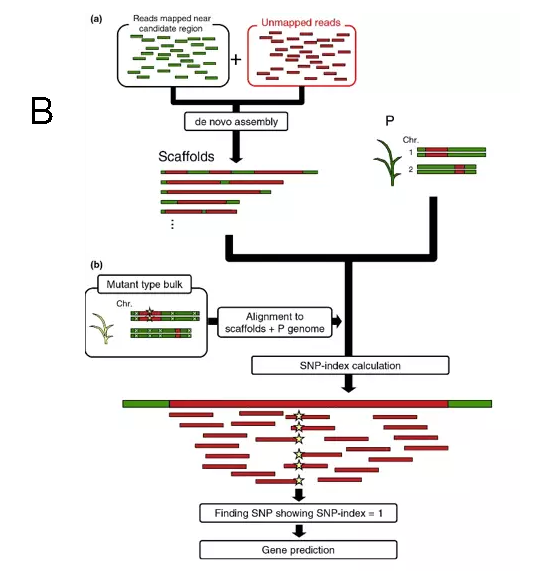
这时候我们将之前比对不上参考基因组的野生型亲本unmapped reads,和位于这个peak区域的野生型亲本mapped reads一起进行denovo组装,得到潜在的新基因,再以此为参考计算SNP-index(图B)。
利用MutMap-Gap方法,这个日本实验室成功鉴定了水稻Hitomebore品系的抗性基因Pii上的一个SNP,而这个抗性基因Pii在Nipponbare参考基因组上是不存在的。因此,MutMap-Gap方法在定位快速变异的基因,如植物抗病基因上,是非常有效的。
说明了MutMap-Gap的原理之后,作者用MutMap-Gap对水稻抗稻瘟基因Pii进行了研究。
那么作者是如何研究的呢?其实一开始作者是用MutMap研究的,发现另外一个变异株的突变位点没有找到,就换了用MutMap-Gap研究了……
MutMap研究思路
据报道, Hitomebore品系中有抗稻瘟基因Pii基因,但是作为水稻参考基因的Nipponbare水稻中并没有Pii基因,于是他们通过重测序得到Hitomebore水稻和Nipponbare水稻的基因序列。并将124,968个Nipponbare基因序列上的SNP位点的核苷酸换成对应的Hitomebore基因序列上的核苷酸,由此构建了一个包含所有Nipponbare SNP位点的Hitomebore“参考序列”。
为鉴别缺失Pii基因功能的Hitomebore突变体,作者将可致稻瘟病变的含AVR-Pii 的033.1喷雾用于经EMS诱变的Hitomebore突变株3033品系中(图a),产生了两种Hitomebore表型突变型Hit5948和Hit6780且都表现出变枯黄病斑叶的突变型(图b)。将两种突变型分别和野生型亲本回交得到的F2都符合3:1性状分离比,说明这两种突变型都是由单隐性基因控制的。

接下来利用MutMap,分别对野生型和突变型的两种F2子代进行混池测序,计算得到SNP-index。
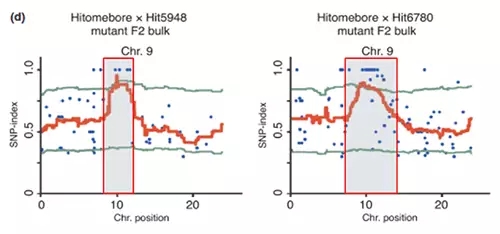
从图中可以看出突变频率接近1的峰值出现在chromosome 9.染色体的:
1、Hit5948的7.88 to 11.98Mb,4个SNP位点的SNP-index为1;
2、Hit6780的7.18 to 13.05Mb,10个SNP位点的SNP-index为1。
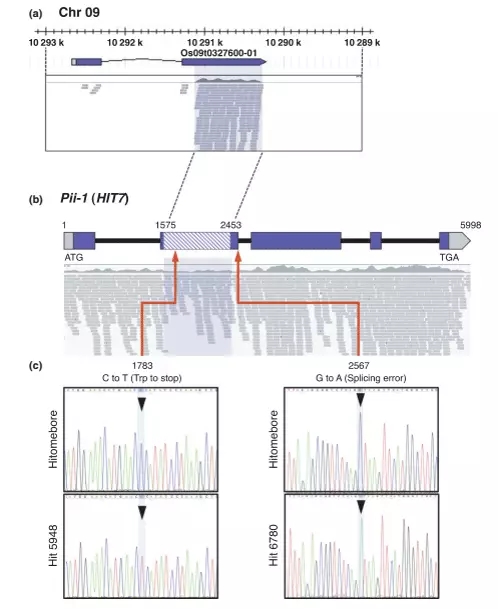
进一步分析发现这些位点都处于Hitomebore的保守植物抗病基因Os09t0327600-01基因上,该基因即为Hitomebore中的Pii基因,相比较Nipponbare中Os09t0327600-01基因因为编码无功能的R蛋白导致抗稻瘟功能的缺失。
图中Hit5948的4个突变位点中,只有位于Os09t0327600-01基因第二个外显子上的SNP-10290916位点上G变成了A,导致色氨酸变为终止密码子,引起表型突变。
Hit6780的10个突变位点中,没有一个发生在Os09t0327600-01基因上的非同义突变,由此,作者推测,这些引起突变的基因,位于Hitomebore特有的基因区域。所以Mutmap未能检测到这些SNPs,需要换个研究思路了。
这个小思路,就是MutMap-Gap!
MutMap-Gap研究思路
首先,将匹配到Hitomebore突变相关的chr09染色体上7.18-13.05Mb区域的4849,550条reads和Hitomebore基因组序列中未匹配到Nipponbare的3358,005条reads进行de novo拼接得到2239条大于1kb的contigs, 由这些contigs组成的852个scaffolds与前文MutMap方法中用到的的Hitomebore“参考基因”合并,得到用于MutMap-Gap的Hitomebore参考基因序列(下图a)。
将Hit6780 F2代混池测序结果与合并得到的Hitomebore参考基因进行比对后发现,与性状突变相关的HIT7基因(后续验证表明,其实就是Pii基因上的一段)上,只有2个位点SNP-index为1,并且导致氨基酸变化(图b,图c),由此作者推测,正是这两个位点的变化导致了Hit6780表型的突变。
为了严谨的科研精神,作者又把突变型Hit5948 的突变基因Os09t0327600-01和突变型Hit6780的突变基因HIT7进行了比对,发现两个基因有极高的相似度。
既然相似度极高,那作者就想了,导致这两种突变型水稻突变的基因,究竟是不是同一个呢?
他们又把Hit5948和Hit6780杂交了,并且对F1代测序,比对Hitomebore参考序列结果发现,突变的2个位点,都位于Pii基因区域!而且杂交的F1代有相同的变枯黄出现病斑叶表型。
结果显示由同一个基因导致的突变。

总 结
MutMap-Gap,就是结合了MutMap和denovo组装的一种基因定位方法,适用于一些:研究品系中特有的但在该物种参考基因中缺失的突变位点定位,所以,如果你通过QTL-seq,MutMap或MutMap+都没有定位到突变基因,那么不妨尝试用MutMap-Gap,可能它们就藏在一些品系特有的基因区域里。
更多基迪奥的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























