如何进行多组学贯穿研究? 返回
宏基因可以了解到微生物群落的组成,多样性与功能等信息,宏转录也是相类似的地了解到这些信息,既然他们作用类似,那还能不能进行组学贯穿研究?贯穿研究又有什么好处?答案就在下面:
1、可以多维度验证物种组成,让群落结构分析更为可靠;
2、基因层面(理论层面)和转录层面(接近实际的发生状态)进行功能研究,功能分析就更为丰富;
3、多组学分析的优势互补,文章思路就更为丰富。
研究思路参考如下:
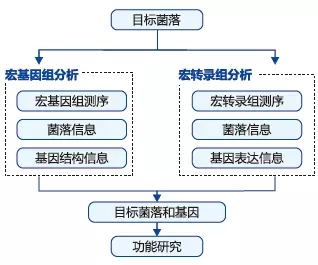
下面我们来探讨一个实际案例看看这种思路如何贯彻。
结肠炎的微生物转录响应机制研究[1]
宿主结肠炎会引起正常肠道微生物变化,但其改变机制仍未清楚。本研究利用宏基因组与宏转录组相结合的策略,研究小鼠(结肠)炎症时期的肠道微生物组成结构和转录活性变化,为后续疾病研究提供重要的参考依据。
主要样本处理
16只小鼠分H.hepaticus处理和无菌处理两组,每组各一半同时注射aIL10R抗体,只有同时经过细菌和抗体处理的小鼠才患有肠炎。14天后取肠道内容物进行宏基因组和宏转录组测序。
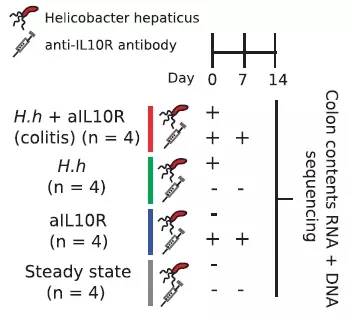
图1 微生物样本分类策略
实验结果
炎症的发生与物种改变息息相关。比较宏基因组与宏转录组两种不同分析手段,虽发现4种样本中所发现的活跃物种并不完全一致,但Firmicute和Bacteriodetes都是它们的优势种。同时,通过宏基因组测序并没有发现患病组的微生物组成以及多样性与其他三个对照组的有明显的改变(p=0.17),但通过宏转录组却显示它们有明显的变化(p=0.045),这表示炎症环境与微生物的转录活性改变有关(图1)。
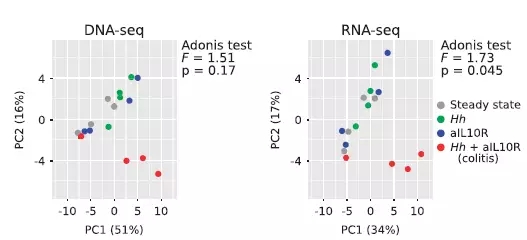
图2 基于宏基因组和宏转录组的样本组成比较
了解完细菌的组成变化情况后,作者进一步了解微生物群落功能与炎症环境的关系。把宏基因组与宏转录组比对到IGC数据库进行eggNOG功能注释,发现宏基因组与宏转录组的数据有很多不同点:
1、宏基因组中功能最多的类别为复制、重组和DNA修复等,而宏转录组数据则显示主要为碳水化合物转运、代谢等功能(图3);
2、只有基于宏转录组的功能聚类能把患病和正常样本分开,而宏基因组则不行。
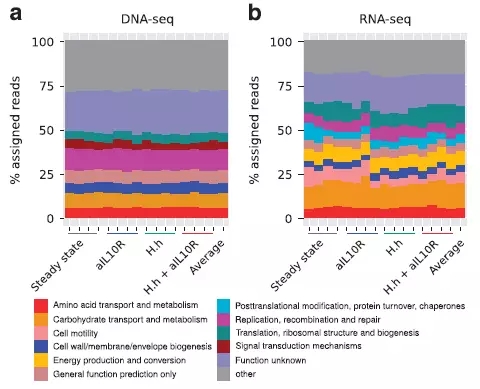
图3 基于宏基因组和宏转录的NOG功能比较
比较患病和正常样本发现,这两类样本在宏基因组数据有1221个差异基因,而宏转录则只有669个。
相比于正常样本,炎症样本细菌中表达增加的NOGs功能包括半胱氨酸合成、过氧化物酶活性、DNA保护和TonB依赖受体等,这些结果与之前的人类肠炎研究结果基本一致。这表明炎症期间细菌中与氧化压力和营养吸收相关的功能基因转录活性改变与炎症环境的应激相关。
同时,比较宏转录组数据和宏基因组数据发现氧化压力拮抗的基因在炎症时期的表达实际变化比宏基因组所预测的大,这也同时表明氧化压力拮抗参与到细菌在炎症时期的应激当中。

图4 与炎症相关的核心NOG功能比较情况
最后,为了了解宿主肠道环境的改变与细菌应激之间的关系,通过宿主转录组数据分析发现宿主肠道产生抗菌物种的基因(如AMPs S100a8,S100a9等)表达有所增加。这些数据与前面细菌样本中含有的人体转录组数据基本吻合(图5),这表示宿主在炎症时期所产生的抗菌物质引起细菌的转录应激改变。

图5 不同处理小鼠的转录改变情况
小结
文章利用宏基因组与宏转录组相结合的情况,在基因和转录层面解答肠道细菌与肠炎的应答机制,从而推测炎症时期肠道的氧化抗菌物质等改变引起肠道共生微生物的转录活性的应激变化。
参考文献:
[1] Ilott, Nicholas Edward, et al. "Defining the microbial transcriptional response to colitis through integrated host and microbiome profiling." The ISME journal(2016).
更多基迪奥的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























