基迪奥合作客户环状RNA高分文章发表! 返回
2017年3月以来Cell Research、Molecular Cell等杂志接连报道了3篇内源性circRNA可以翻译蛋白质的研究,但对翻译的新蛋白生物学功能和分子调控机制仍不清楚。
8月29日,中山大学附属第一医院神经外科张弩副教授团队在国际顶级学术期刊美国国家癌症研究所杂志JNCI(Journal of the National Cancer Institute,IF2016 = 12.589)发表题为“Novel Role of FBXW7 Circular RNA in Repressing Glioma Tumorigenesis”的论文,在国际上首次证实circ-FBXW7可以翻译一种抑制胶质瘤的全新蛋白质,并用功能学实验证明了新蛋白的分子机制,在肿瘤circRNA研究中具有里程碑意义!
基迪奥有幸参与该项目,配合完成了circRNA的高通量测序和生物信息学分析,准确鉴定和筛选了神经胶质瘤相关的circRNAs,并且出色完成老师的个性化分析和定制化图表的绘制,为后续功能学验证打好基础。
那么作为一篇以测序为基础,多种功能学实验结合的高分文章,究竟有哪些值得借鉴的思路和方法呢?下面我们为大家一一解读,重点讲解以下三点:
1、如何筛选和确定目标circRNA分子?
2、如何推测和证实circRNA翻译蛋白质?
3、如何研究circRNA翻译蛋白质的生物学机制?


发表期刊:《JNCI J Natl Cancer Inst》 [1]
影响因子:12.589
研究背景
最近研究表明circRNA在发育,基因调控和癌症发生发展过程中发挥着重要作用,但circRNA直接编码蛋白发挥生物学功能的报道还很少,相关研究还有很大的空白。
研究思路
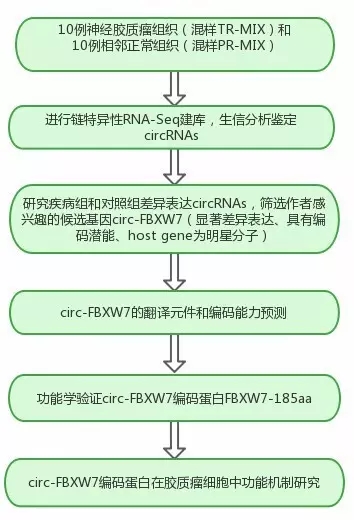
研究结果
1、circRNA高通量测序和差异表达研究
对脑胶质瘤临床组织标本进行circRNA深度测序及生物信息学分析挖掘,得到下机数据后,首先将会对其进行过滤,得到HQ Clean Reads。每个样品的 HQ Clean Reads利用Botiwe2和TopHat分别与核糖体序列和参考基因组比对,从比对结果中提取Unmapped Reads,然后截取每一条Unmapped Reads的两端(默认20bp),得到Anchors Reads。用Anchors Reads再一次比对到基因组上,将得到的比对结果提交给find_circ软件鉴定出环状RNA。
总共发现了31145个circRNAs,其中6442个已经收录在circbase数据库中。大多数circRNAs(18399个)来源与外显子区域,其它来源于内含子,5’-UTR, 3’-UTR区域或者antisense sequences。绝大多数circRNAs的长度小于1500bp,其中小于500bp的数量最多。癌和癌旁中circRNAs的染色体分布没有明显差异,但是癌组织中circRNA整体表达丰度低于正常组(图1)。
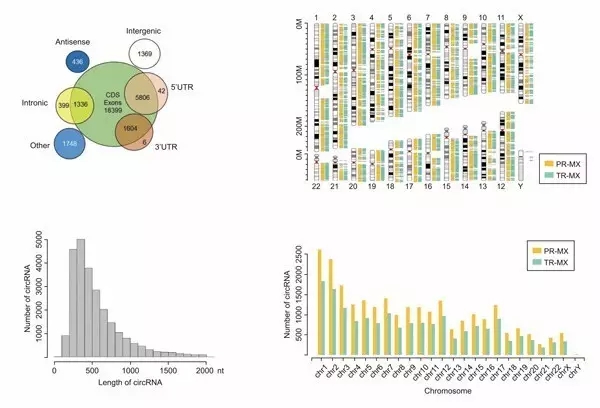
图1. CircRNAs测序信息统计
2、筛选和确定目标circ-FBXW7分子
首先,通过上述circRNA深度测序及生物信息学分析挖掘,发现了一些在脑组织以及癌旁组织中有明显表达差异的circRNAs。
其次,作者关注了著名的抑癌基因FBXW7基因的第3、4外显子环化后形成一个620 nt的环状RNA,并命名为circ-FBXW7。
最后,作者根据circRNADb在线数据库检索了一下,发现此数据库收录了FBXW7基因对应的一个circRNA跨越基因组长度1227bp的分子,剪接的成熟体circRNA是620nt,和本研究报道的吻合,暗示着circ-FBXW7可能具备蛋白编码的潜能(图2)。综合以上特点,作者将研究目标锁定为circ-FBXW7,是整篇文章的基石。
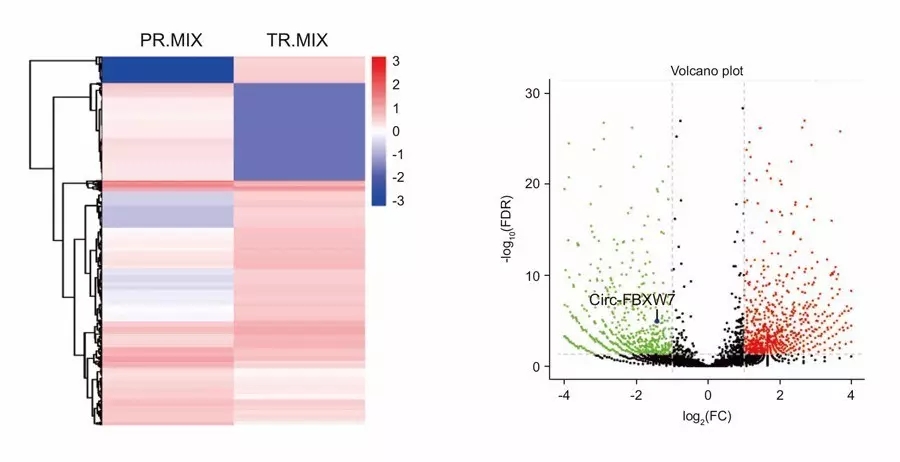
图2. CircRNA差异表达和候选基因的筛选
3、功能学验证circ-FBXW7的环状大小和结构
为了证明circ-FBXW7来源于host gene的3,4外显子,作者设计特异性引物,利用qPCR发现cDNA中有环状预期的片段,并且抵抗RNase R的消化;此外,gDNA没有预期的片段,证明circ-FBXW7为环状RNA的可靠性。Sanger测序验证环化接头位点的序列;此外还通过构建circRNA表达载体验证了circ-FBXW7长度为620nt,不同于circbase中的1227nt,作者推测这是circRNA内部存在可变剪切有关(图3)。
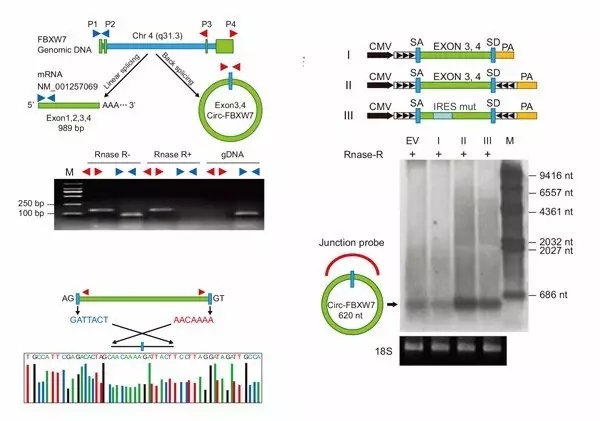
图3. Circ-FBXW7的环状结构的功能性验证
4、Circ-FBXW7的编码能力预测和功能性验证
作者分析Circ-FBXW7的翻译元件,首先,发现其存在一个编码185 aa的开放阅读框(ORF),并且具有物种间保守性暗示ORF的编码潜力;其次,包含核糖体进入位点(IRES)序列,预示着具备基本的翻译元件。 接着用双荧光报告实验证明Circ-FBXW7中的IRES的相关活性,具有引导5’帽子非依赖性翻译的能力;Flag标签表达载体证明Circ-FBXW7能够在人类细胞中翻译出相关蛋白;最后用LC-MS质谱技术证明翻译的蛋白约为21KD,并命名为FBXW7-185aa。

图4. Circ-FBXW7的编码能力预测和功能性验证
5、circRNA翻译的FBXW7-185aa蛋白在肿瘤中的生物学活性和分子机制
通过细胞功能试验,作者发现FBXW7-185 aa蛋白能够明显抑制肿瘤细胞的增值。为了证明是FBXW7-185 aa蛋白发挥功能而非Circ-FBXW7的作用,作者突变了IRES元件作为对照。IRES序列突变后,circRNA可以形成但是翻译蛋白质就受到很大影响,这就证实FBXW7-185 aa蛋白而不是RNA发挥着直接的作用,也是文章的亮点。
根据已有研究报道,FBXW7基因作为一个E3泛素连接酶,通过泛素化途径调控一大批癌基因的稳定性,包括著名的原癌基因c-Myc。FBXW7产生的蛋白FBXW7a能够降低了c-Myc的稳定性。
作者首先证明circRNA翻译的小蛋白FBXW7-185aa也能明显降低c-Myc蛋白的表达,并证明FBXW7-185aa影响c-Myc的半衰期。通过IP和pulldown实验证实FBXW7-185aa蛋白可以竞争性结合USP28,从而促进全长FBXW7产生的蛋白对c-Myc蛋白的泛素化降解,从而参与调控肿瘤生物学过程。因此,作者发现了FBXW7产生的蛋白和FBXW7-185aa协同作用调控c-Myc的有趣的调控模式。
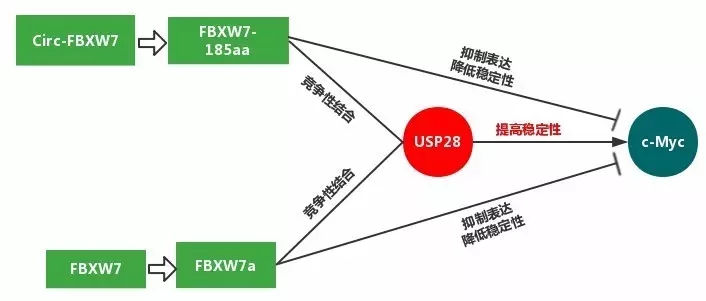
图5. FBXW7-185aa蛋白功能机制模式图
研究总结
本文是一篇典型的高通量测序筛查目的基因,再通过完整的功能学实验验证的高分医学文章,其关键点至少有以下几个方面:
1、高通量测序的为研究者筛选了与胶质瘤显著相关的circRNAs,这些差异表达的circRNAs可能都以各种方式参与了胶质瘤的发生发展过程,为研究者提供了重要的候选分子储备。因此,高通量测序为临床分子机制研究提供了可靠的和丰富的组学候选分子。
2、作者在一批差异表达基因中毅然选择了来源于抑癌明星分子FBXW7的circRNA作为研究对象,为整篇研究打好了基础。首先,来源基因的功能可能直接影响形成的circRNA生物学特性;其次,候选circRNA在疾病组和对照组间显著差异表达,很大几率发挥着生物学功能;最后,候选circ-FBXW7含有完整的ORF和IRES翻译元件,其编码能力研究也是circRNA研究的重要方向。因此,基于以上考虑作者明智的选择了目的基因,可以说是整个研究的关键。
3、circ-FBXW7的结构特性,根据已报道的circRNA翻译文章,并非所有具备翻译能力的circRNA都具有IRES,有些没有IRES序列在m6A修饰下驱动翻译[2],有些具有类IRES功能的短序列(如circMbl cUTR)驱动非帽翻译[3],候选circ-FBXW7含有完整的ORF和IRES翻译元件一方面更具有翻译潜力,另一方面为IRES突变等功能学实验带来结构上的便利。
4、circRNA的翻译能力的研究是前沿和新颖的,作者通过大量的体内和体外功能性实验如免疫学和蛋白质谱等,得出了一系列直接的数据,证明了circRNA编码的蛋白和其生物学功能。
5、作为肿瘤circRNA的第一篇蛋白翻译的文章,它的意义在于揭示了circRNA除了通过miRNA sponge功能参与癌症功能外,circRNA还可以通过直接编码的蛋白来影响癌症的生物学进程,打破了将circRNA定义为“非编码RNA”的传统认识,拓展了circRNA在肿瘤学中的研究领域。
基迪奥公司为客户提供了优质全面的高通量测序和个性化生物信息服务,具有丰富的circRNA项目经验,能为您提供:专业的方案设计——个性化方案服务。丰富的项目经验——成熟的circRNA高通量研究和生物信息学分析,包括对组学circRNA数据的ORF预测等翻译功能的研究。可直接发表的内容展示——针对客户定制化生物信息分析和个性化图表绘制服务。
参考文献:
[1] Yibing Yang, Xinya Gao, Maolei Zhang, Sheng Yan, Chengjun Sun, Feizhe Xiao, Nunu Huang, Xuesong Yang, Kun Zhao, Huangkai Zhou, Suyun Huang, Bo Xie, Nu Zhang (2017) Novel Role of FBX W7 Circular RNA in Repressing Glioma Tumorigenesis. JNCI J Natl Cancer Inst. doi: 10.1093/jnci/djx166
[2] Yang Y, Fan X, Mao M, Song X, Wu P, Zhang Y, Jin Y, Yang Y, Chen L, Wang Y, Wong C C, Xiao X and Wang Z (2017). extensive translation of circular RNAs driven by N6-methyladenosine. Cell Res. doi: 10.1038/cr.2017.31
[3] Pamudurti N R, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perez-Hernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N and Kadener S (2017). Translation of CircRNAs. Mol Cell. doi.org/10.1016/j.molcel.2017.02.021
更多的原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























