如何绘制序列保守性Logo图? 返回
在一些高分文章中经常会看到Logo图,看着非常赏心悦目,如下图:
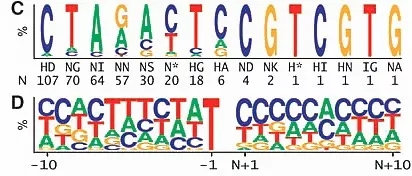
(Science,2009)
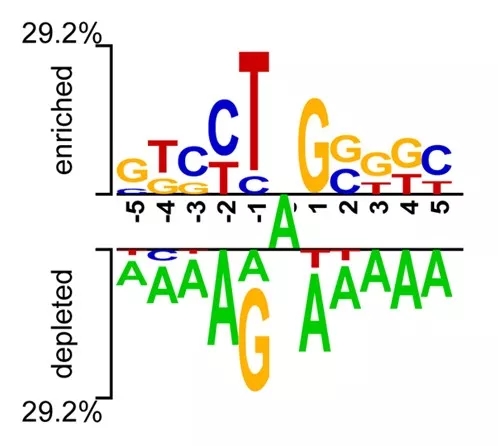
(PLoS Genet,2014)
那么类似这样的Logo图该怎么绘制呢?
1、序列准备
首先,将目标序列进行多序列比对,这里使用MEGA的Alinment功能,方法选内置的Align by ClustalW,如下图。
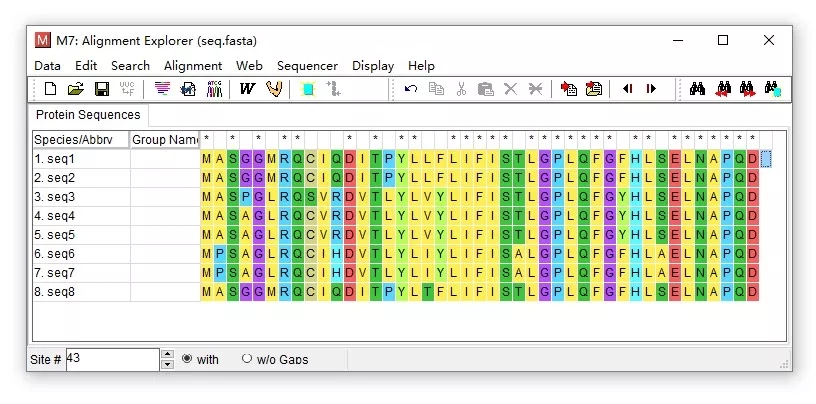
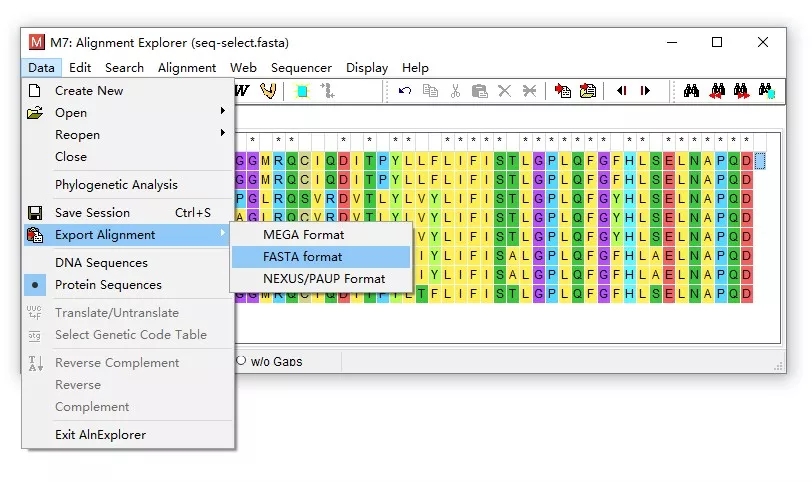
“对齐”后,将首尾两端“裁齐”,为了方便展示这里把序列裁剪得很短,仅使用序列前段的四十多个氨基酸。裁剪方法:框选不要的序列部分,按Delete键即可。然后将裁剪后的序列导出为fasta格式,方法见下图。
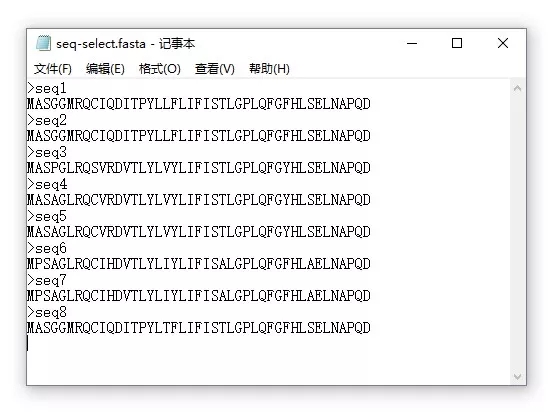
保存为fasta格式后的序列用记事本打开是这样子的:
2、绘制方法
绘制方法有很多,这里介绍一个在线工具:WebLogo (http://weblogo.berkeley.edu/),目前有两个版本,如下图,最新版本是3.6。

使用方法很简单,和几乎所有的在线工具一样(以2.8的版本为例),通过选择文件 或 直接粘贴的方法上传序列,如下图。
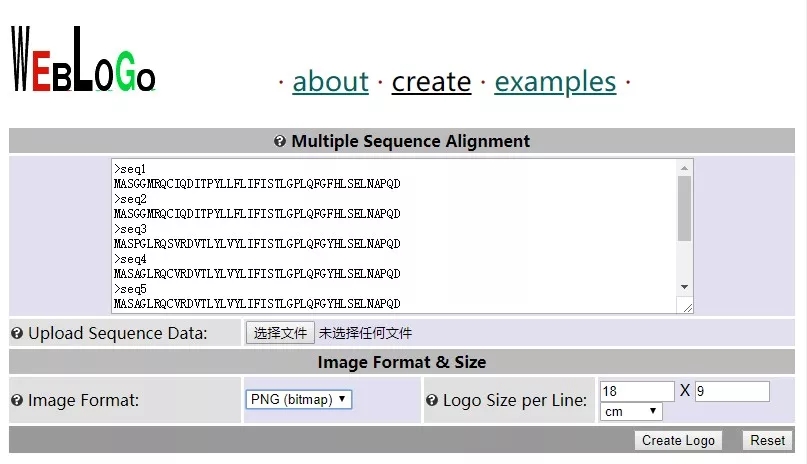
图片的格式可选svg、PDF等矢量格式,这里用PNG格式,dpi选默认的 96(因为我这里用仅用于网络传播),图片的尺寸默认即可。
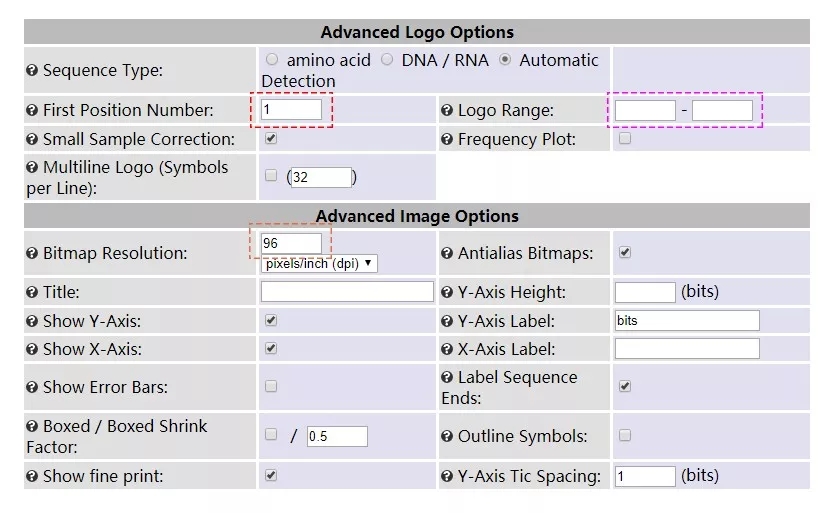
所需要注意的是序列显示范围的设置,默认是显示所有序列的字母,如需要仅显示其中的一小段,可设置显示字母范围(如下图洋红色虚线框所示),红色线框所示的是设置横轴的起始数字。
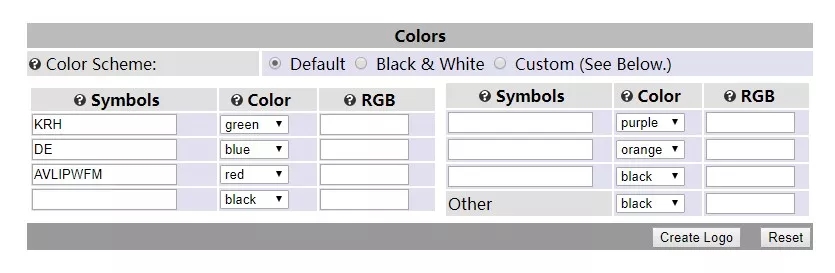
另外,字母的颜色也可以自定义,如下图。
绘制效果见下图:
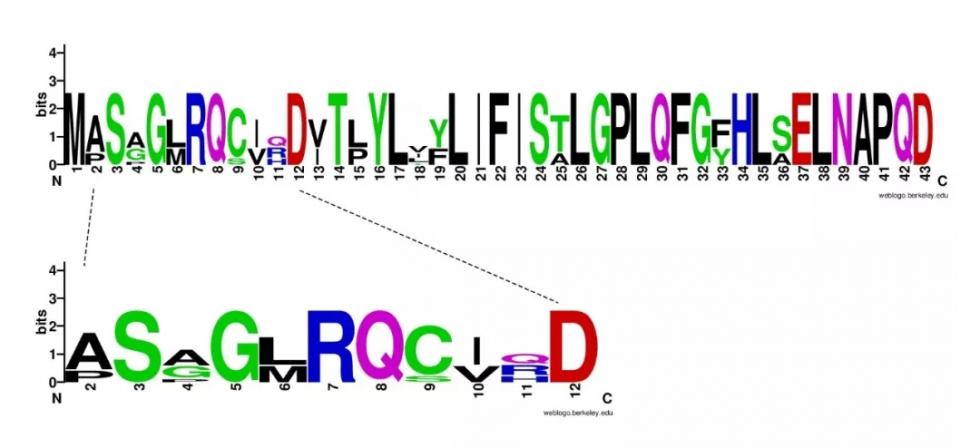
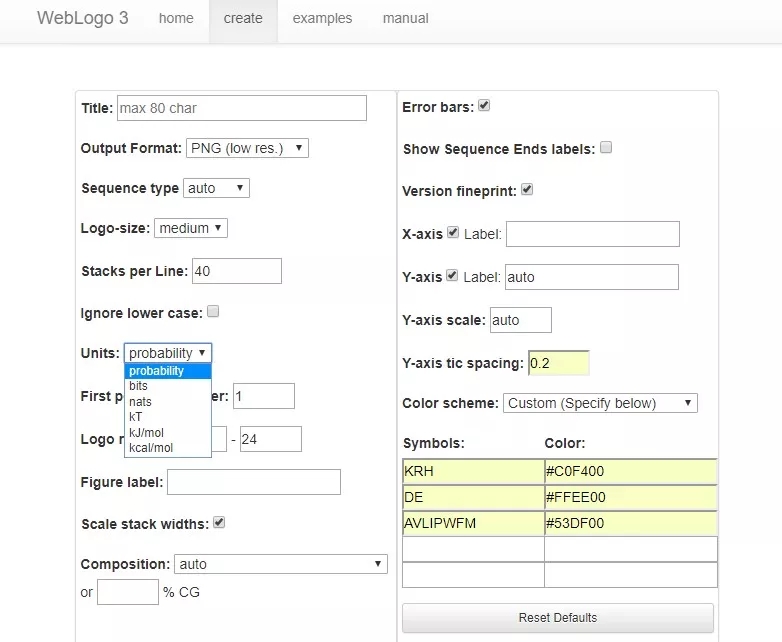
如果想绘制以百分比为单位的Logo图,可用3.6版本(2.8 版本的y轴显示貌似有问题),所用方法大同小异,单位选 probability 即可,颜色可自定义,如下图。
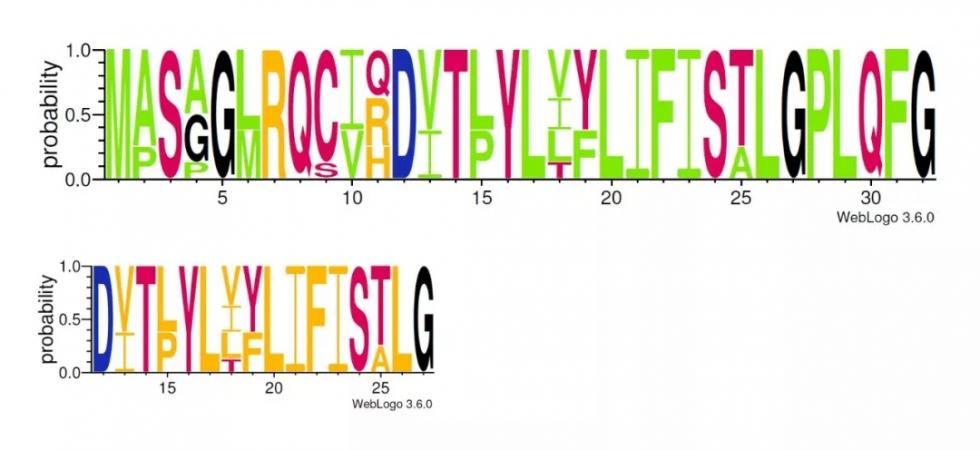
绘制的效果如下:
3、Logo图的解读
序列logo图由TomSchneider 和Mike Stephens发明,用来分析和展示序列模式的保守性。
图中的每个字母的高度与该位置的相应碱基或氨基酸残基的出现频率成正比,常以bits为单位。每个位置的字母按照保守性从大到小排列,可以方便的从顶端的字母识别保守序列,例如下图(B)CAP的保守序列是“AA-TGTGA------ TCACA-TT”。
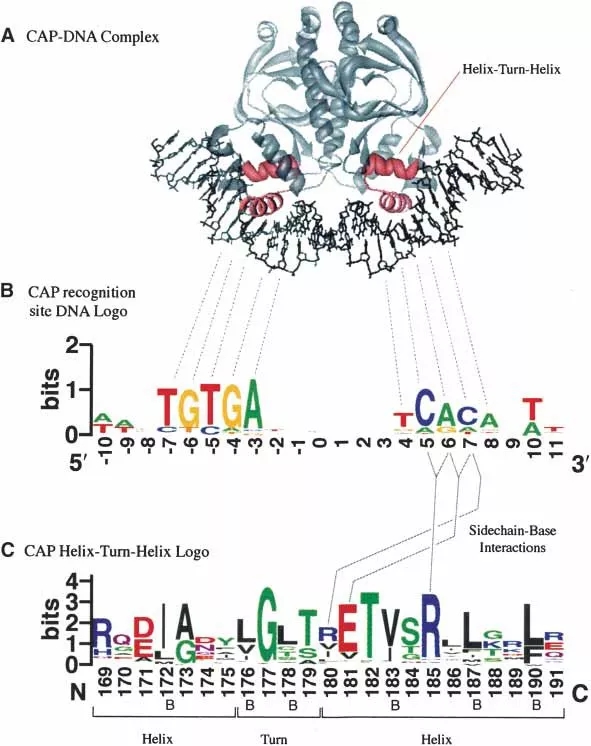
(GenomeResearch,2004)
纵坐标的单位常见有两种,一种是百分比,另一种是bits。前一种好理解,每个字母的出现频率;对于后一种,可参考下面的公式:
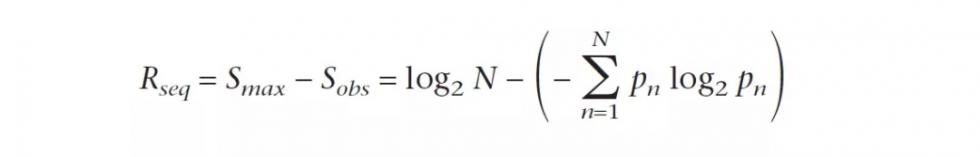
这里,pn是相应位置n上相应字符出现频率,N是不同字符的总数量(核酸为4,蛋白质为20)。因此,对于图中的y轴数值就不难理解,核酸序列的最大值为log2 4 = 2bits,蛋白序列为log2 20≈4.32 bits。
参考文献
Chen J Y, Peng Z, Zhang R, et al. RNA editomein rhesus macaque shaped by purifying selection[J]. PLoS genetics, 2014, 10(4):e1004274.
Steczkiewicz K, Muszewska A, Knizewski L, etal. Sequence, structure and functional diversity of PD-(D/E) XKphosphodiesterase superfamily[J]. Nucleic acids research, 2012, 40(15):7016-7045.
Moscou M J, Bogdanove A J. A simple ciphergoverns DNA recognition by TAL effectors[J]. Science, 2009, 326(5959):1501-1501.
Crooks GE, Hon G, Chandonia JM, Brenner SEWebLogo: A sequence logo generator, Genome Research, 14:1188-1190, (2004)
Schneider TD, Stephens RM. 1990. SequenceLogos: A New Way to Display Consensus Sequences. Nucleic Acids Res.18:6097-6100
更多的动态,可继续关注我们网站,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























