组内/组间相关性分析工具使用教程 返回
基因表达的高低有可能影响着代谢物的种类和丰度;
温度和pH等环境因素无时无刻不影响着微生物的群落结构;
不同组学数据之间往往存在关联,如何从这些复杂的数据变化寻找到本质的关联规律?这需要特定的数学模型去挖掘,在众多模型中,最常用的莫过于相关性分析。
为方便老师们挖掘数据之间的相关性,此次在云平台新增“相关性分析工具”。工具提供最常用的基于pearson和spearman两种方法的分析策略(两种分析方法的差异,如下链接)。
为实现对同一组学内和两个不同组学之间元素的关联信息挖掘,我们同时提供组内相关性和组间相关性两个平行的分析工具,以尽可能满足更多的科研需求。
工具网址
组内相关性分析 http://www.omicshare.com/tools/Home/Soft/ica
组间相关性分析 http://www.omicshare.com/tools/Home/Soft/ica
操作步骤
1、进入工具
点击以上链接,或登录Omicshare 云平台 → “我的软件”→ 搜索“组内相关性分析”/“组间相关性分析”。

图1 组内相关性分析图标 图2 组间相关性分析图标
2、输入文件
图3 输入文件界面(组内)
图4 输入文件界面(组间)
组内相关性分析只需输入一个文件,包含分析数据及表头信息,且为tab-delimited所保存的txt格式,如下图基因表达量表格:
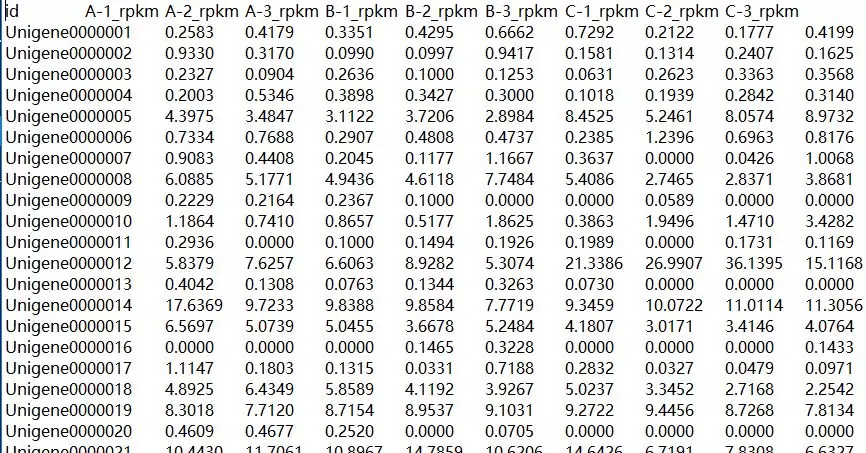
图5 输入文件示例(组内)
组间相关性分析必须输入两个表格,可以为两个组学数据,例如基因表达量表、代谢物丰度表、微生物丰度表等。也可以是表型数据,例如升高体重等生理指标。
表格需要带表头和列名,每一列应代表样本,行代表各种元素,例如OTU,基因。
表格必需为tab-delimited所保存的txt格式,如下图基因表达量与代谢物丰度表格:
文件1,12个样本代谢物丰度表。
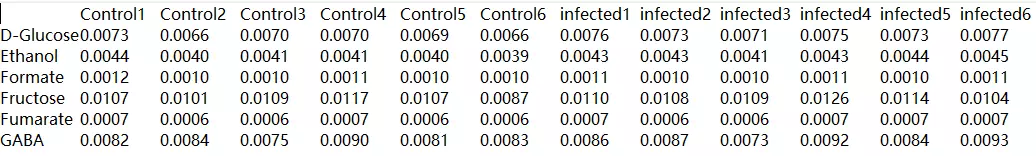
图6输入文件1示例(组间)
文件2,12个样本基因表达量表。
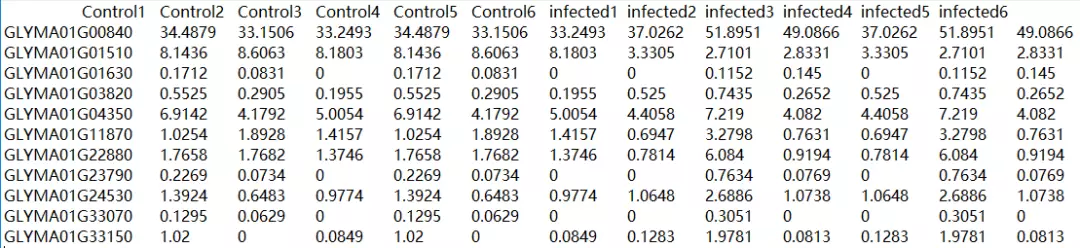
图7输入文件2示例(组间)
3、设置参数
用户可根据数据量大小等选择参数。组内、组间相关性参数如下图:
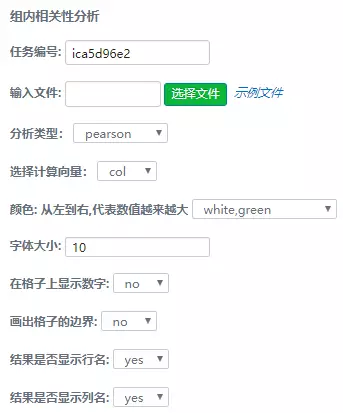
图8 参数界面(组内)
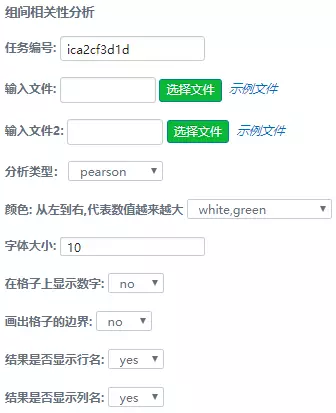
图9参数界面(组间)
① 分析类型:可选择计算pearson系数或spearman系数。
② (组内相关性分析)计算向量:选择计算表格中每两行(row)或每两列(col)数据的相关系数,如示例文件中,若选择“col”,则计算样本之间的相关性,可用于评估样本分组差异、样本重复性等;若选择“row”,则计算基因之间的相关性,可用于评估基因的关联特征等。
③ 颜色:可根据数据量大小、相关系数波动范围等选择2种或3种颜色。可选择候选颜色或手动输入。
④ 字体大小:可调整横纵坐标名的字体大小。
⑤ 在格子上显示数字。
⑥ 画出格子边界。
⑦ 结果是否显示行名。
⑧ 结果是否显示列名。
4、提交
提交后界面如下
图10任务提交界面
5、查看结果
待倒计时结束,点击“刷新任务状态”,如下图红框位置。
图11任务刷新界面
刷新后可查看结果,如下图:
图12结果查看界面
点击“文件预览”,即查看相关性热图。
点击“下载文件”,可下载系数表格及热图,示例如下。
① 组内相关性分析输出1个表格内每两行(或列)数据的相关系数表格及热图。
结果示例(平行样本重复性评估):
all.cor_heatmap.png/pdf:两种格式的热图文件
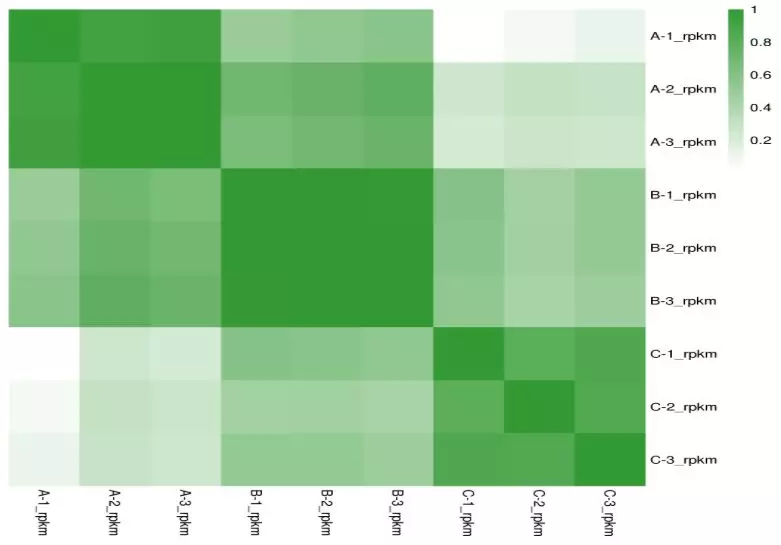
图13 相关系数热图(组内)
横纵坐标都表示样品名称,每3个平行样品之间相关性最高(对角线深色区域),表明平行样本的重复性很好,样本分组间差异较大(对角线以外浅色区域)。
all.cor.matrix.xls:相关系数矩阵,绘制热图的数据格式,如下
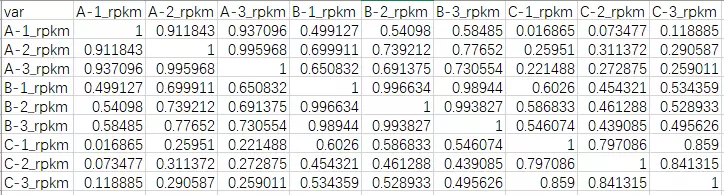
图14 相关系数表(组内)
all.pvalue.matrix.xls:相关显著性p值矩阵,如下
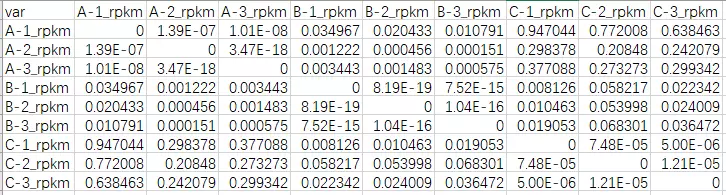
图15 相关显著性p值表(组内)
all.cor_pvalue.list.xls:相关系数及p值列表,是方便大家使用云平台工具绘制网络图的数据格式,如下
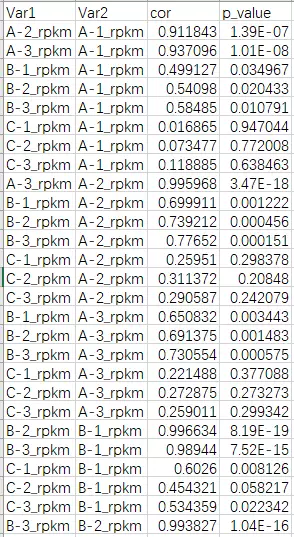
图16 相关系数和显著性p值表(组内)
② 组间相关性分析输出2个表格之间每两行数据的相关系数和热图。
结果示列(转录组和代谢组的关联分析):
all.cor_heatmap.png/pdf:两种格式的热图文件
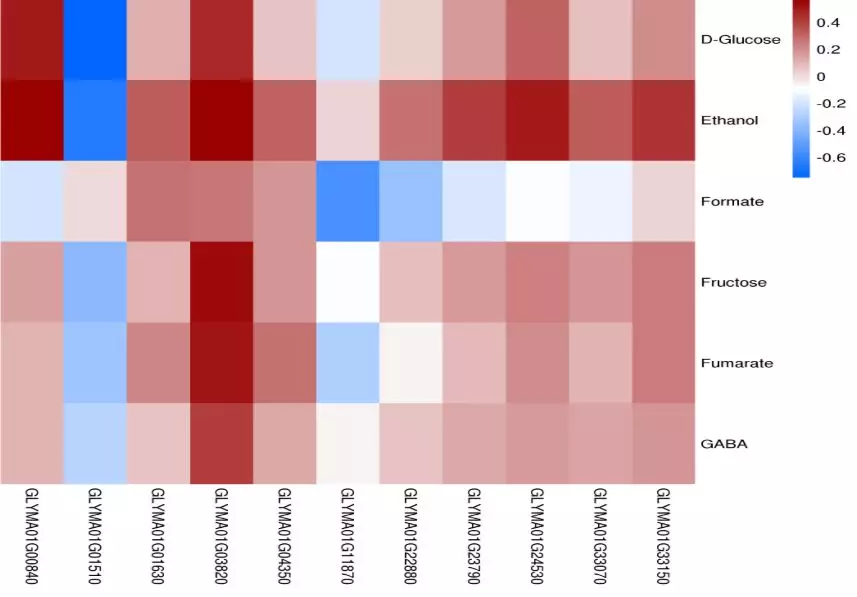
图17 相关系数热图(组间)
图中横坐标为基因名称,纵坐标为代谢物名称,展示了数据中某些基因与代谢物之间密切的相关性(深红和深蓝区域),为下一步的深入分析(如网络图)奠定基础。
all.cor.matrix.xls:相关系数矩阵,绘制热图的数据格式,如下

图18 相关系数表(组间)
数值即为对应行代谢物与对应列基因的相关系数。
all.pvalue.matrix.xls:相关显著性p值矩阵,如下
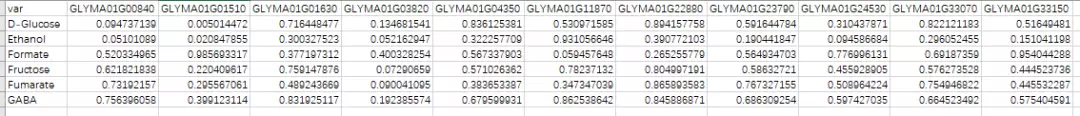
图19 相关显著性p值表(组间)
all.cor_pvalue.list.xls:相关系数及p值列表,是方便大家使用云平台工具绘制网络图的数据格式,如下:
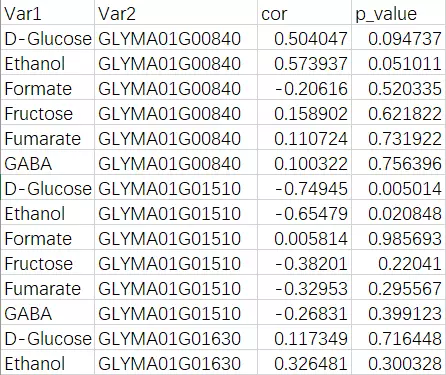
图20 相关系数和显著性p值表(组间)
附:常用判断标准
相关显著性p <0.05,代表显著相关;p <0.01,代表极显著相关。
相关性强弱判断:
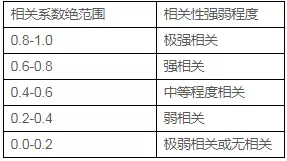
更多的动态,可继续关注我们网站,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!



























