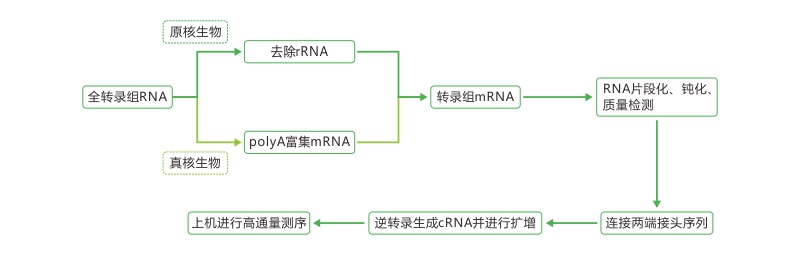
|
参考文献
[1] Liu C H, Liu Y, Shao X H, et al. Comparative Analyses of the Transcriptome and Proteome of Comte de Paris and Smooth Cayenne to Improve the Understanding of Ethephon-Induced Floral Transition in Pineapple[J]. Cellular Physiology and Biochemistry, 2018, 50(6): 2139-2156.
[2] Yuan K, Xie X, Wang X, et al. Transcriptional response of Mycobacterium sp. strain A1-PYR to multiple polycyclic aromatic hydrocarbon contaminations[J]. Environmental Pollution, 2018, 243: 824-832.
[3] Zheng T, Qi P F, Cao Y L, et al. Mechanisms of wheat (Triticum aestivum) grain storage proteins in response to nitrogen application and its impacts on processing quality[J]. Scientific reports, 2018, 8(1): 11928.
[4] Guo J, Qi J, He K, et al. The Asian corn borer Ostrinia furnacalis feeding increases the direct and indirect defense of mid‐whorl stage commercial maize in the field[J]. Plant biotechnology journal, 2018.
[5] Li N, Yang L, Qi X K, et al. BET bromodomain inhibitor JQ1 preferentially suppresses EBV-positive nasopharyngeal carcinoma cells partially through repressing c-Myc[J]. Cell death & disease, 2018, 9(7): 761.
|