文献解读 | RNA-seq研究植物抗病的思路 返回
在我们处理过的RNA-seq类项目中,植物抗病项目占据了大部分。那么RNA-seq数据到底能否助力植物抗病研究?为此,基迪奥的周老师给大家解读一篇植物抗病研究的SCI论文。

这篇文章题目是“利用RNA-seq分析大豆近等基因系抗病株系和感病株系的基因表达模式差异”。
第一部分:背景
1、什么是近等基因系
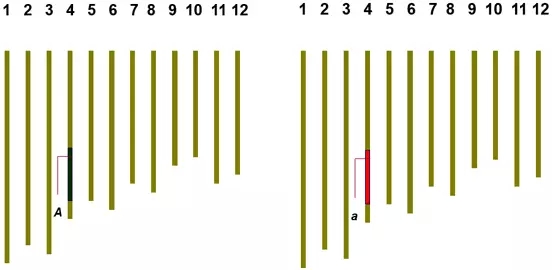
近等基因系,就是一系列遗传背景几乎完全相同,只有一段染色体不同的材料。这类材料一般通过不断地回交和自交来获得。理论上,这类材料的性状差异,只由于一条基因片段的差异导致(上图中的A和a)。因此,它是基因等位和功能研究的理想材料。
2、文章主体结构
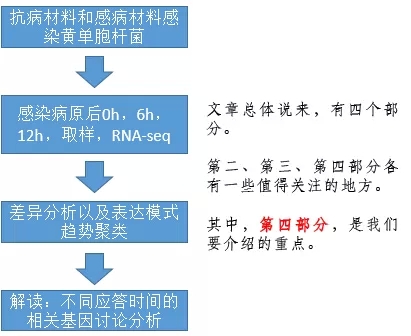
第二部分:感染的时间
这里的0h,6h,12h对应早、中、晚期,这个设计还是非常有技术含量的。
例如:病原感染后,可能会有一定时间的休眠期,到底什么时候开始增殖,可能需要提前做Q-PCR来确定。
第三部分:差异分析以及表达模式聚类
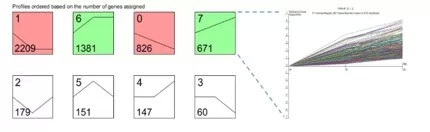
将差异基因分为10种不同的表达模式,这将作为后续分析讨论的基础。
第四部分 :数据的解读
先看下面的通路图:
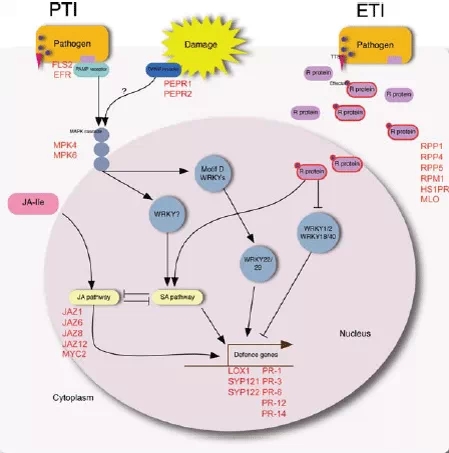
这张图涉及到PTI(广谱性防御)、ETI(特异性防御),以及机体受到损伤(Damage)导致的调控变化。
第四部分:不同时间点的调控变化
作者主要先对3类基因分开进行讨论:
1、PTI相关基因,也就是病原广谱性防御相关的受体蛋白(识别病原蛋白)。
2、ETI相关的基因,识别特异的病原蛋白。
3、激素调控相关的基因。
分别统计后,不同的时间点(0h,6h,12h)差异基因的出现有一定规律(如下):
早期(0h):PTI相关基因,钙离子流蛋白,氧化爆发相关基因。这类基因主要参与识别微生物蛋白(外源蛋白)。
中期(6h):主要涉及蛋白激酶的变化(MAPK等)。这类基因主要将病原蛋白识别信号传递到下游的细胞防御通路。
晚期(12h):主要涉及水杨酸累积(SA)等相关的基因。这些基因将调控细胞发送形态变化,从而起到对病原防御的作用。
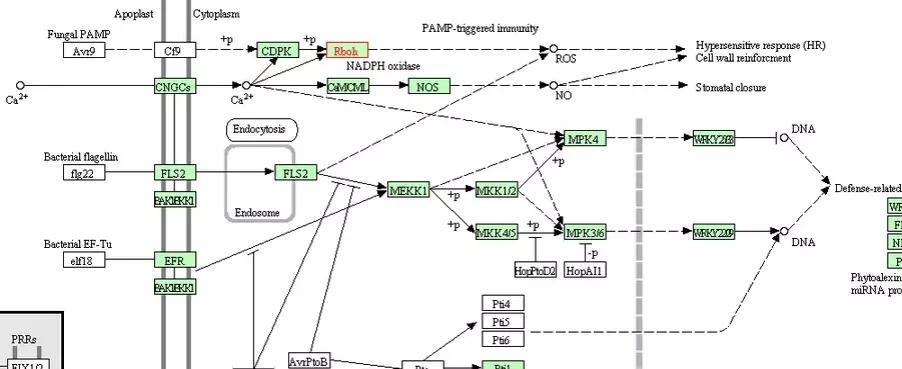
重点提醒:KEGG的通路包含了大部分此类重要的基因(如上图)。而这些信息在我们的项目分析结果中都可以找到,老师只要找出这些信息并解读就可以了。
第四部分:抗病基因(R基因)的差异表达
以上涉及到特异性防御的基因调控变化,都和抗病基因(R基因)识别微生物蛋白有关。
那么R基因是否会差异表达?

文章发现3个相关的候选基因没有表达差异变化。原因可能有很多。不过作者解释到,还可能是由于取叶片的时候取到了未感染的部位,稀释了差异倍数有关。
小结:
1)通过合理设计多个时间点,结合对RNA-seq结果的解读,可以很好描述微观水平基因表达的动态调控过程;
2)仅仅利用RNA-seq来定位R基因是很困难的。但通过合理的实验设计,再结合其他技术,如QTL定位、功能验证等,推动R基因定位。
关于植物抗病中期、晚期基因 转基因研究的文章解读,详情点击:http://www.genedenovo.com/news/251.html
更多基迪奥精彩原创文章,可继续关注我们网站动态发布,同时关注基迪奥微信~扫一扫添加基迪奥好友~随时随地关注行业动态!


























